Capítulo 11 Caso de estudio
El objetivo del siguiente proceso de anonimización es ilustrar el proceso SDC establecido en la GUÍA PARA EL CONTROL DE DIVULGACIÓN ESTADÍSTICA EN MICRODATOS propuesta por la Mesa de Anonimización INE. Los datos utilizados corresponden a una base de datos sintética que se basa en el diseño de la 17a ENUSC 2020 (Instituto Nacional de Estadísticas de Chile 2021a).
El proceso SDC aquí implementado pretender generar un archivo de datos PUF (del inglés, Public Use File), que pueda ponerse a disposición de forma pública y para cualquier usuario que lo requiera. Para ello, el proceso SDC aplicado apunta al logro de los umbrales de riesgo establecido por la GUÍA PARA EL CONTROL DE DIVULGACIÓN ESTADÍSTICA EN MICRODATOS para encuestas de hogares (Instituto Nacional de Estadísticas de Chile 2021b). Simultáneamente, el ejercicio busca mantener la utilidad de los datos, intentando minimizar la intervención de los mismos para que los usuarios puedan replicar las estadísticas priorizadas por esta encuesta.
Los umbrales de riesgo establecidos son los siguientes:
RIESGO GLOBAL
- Riesgo global inferior al 10%
RIESGOS INDIVIDUALES
- Hasta 20% de observaciones con riesgo individual mayor al 1%
- Hasta 15% de observaciones con riesgo individual mayor al 5%
- 0% de observaciones con riesgo individual mayor al 25%
K-ANONIMATO
- 0% de observaciones violando k = 2
- Hasta 5% de observaciones violando k = 3
- Hasta 10% de observaciones violando k = 5
Nota sobre el cálculo del K-ANONIMATO: A pesar de que en el ejemplo actual se realiza el cálculo de k-anonimato con la función
Kanon_violations(), se recomienda seguir el estándar del INE, que consiste en calcular el k-anonimato sin ponderadores utilizando el comandoprint(sdcObject). En este contexto, ‘sdcObject’ representa el objeto sobre el cual es necesario evaluar el riesgo.
A continuación, la guía presenta paso a paso como proceder con este conjunto de datos para lograr el cumplimiento de estos umbrales de riesgo.
11.1 Paso Uno: Definiciones previas al proceso de anonimización
11.1.1 Definición del equipo de trabajo
En esta primera etapa, se define el equipo de trabajo, el cual debe estar compuesto al menos por un encargado temático, encargado de los criterios para la selección de variables claves y definición de escenarios, y un analista de anonimización, encargando de la implementación de la misma a través de un script como el aquí expuesto.
11.1.2 Insumos y/o productos necesarios para la ejecución del proceso
En esta etapa se describe la metodología del producto estadístico, la revisión temática de la base de datos, su caracterización y clasificación de variables, la infraestructura con que se cuenta para el proceso, entre otros elementos que se especifican en el documento adjunto.
11.1.2.1 Archivo de Base de datos
Antes de cargar los datos, declaramos el directorio de trabajo donde se ubican los archivos con que trabajaremos.
Bloque 11.1 Indicar el directorio de trabajo
yourdirectory<-"/simulacion"
setwd(yourdirectory)Se trabaja con la base bruta de la ENUSC, que contiene todas las variables previo a la innominación (en este caso es una base de datos sintética).
Bloque 11.2 Indicar el nombre del archivo de datos
fname <- "data/BD_ENUSC_SINTETICA_ETIQUETADA.sav"
file <- haven::read_sav(fname)11.1.2.2 Caracterización de la base de datos
Primero revisamos las dimensiones del archivo, es decir, cuantos registros y variables contiene. La base contiene 47.344 registros y 73 variables. Luego, revisamos el nombre de las variables contenido en la base de datos para verificar que se ajusta a la base con que se requiere trabajar.
Bloque 11.3 Inspeccionar las dimensiones del conjunto de datos y los nombres de las columnas
dim(file)## [1] 47344 73names(file)## [1] "enc_idr" "rph_ID" "enc_id"
## [4] "enc_region" "enc_rpc" "enc_distrito"
## [7] "enc_zona" "enc_manzana" "enc_vivienda"
## [10] "IDC" "enc_letraKish" "enc_direccion"
## [13] "enc_numero" "enc_codfono" "enc_fono"
## [16] "enc_Nombre_ID" "Enc_Fono_ID" "Enc_Correo_ID"
## [19] "enc_Nombre_K" "enc_Edad_K" "Enc_Fono_K"
## [22] "Enc_Correo_K" "FECHA" "IH_residencia_habitual"
## [25] "Hora_inicio_rph" "rph_numeroLinea" "rph_nombrepila"
## [28] "rph_parentesco" "rph_dianacimiento" "rph_mesnacimiento"
## [31] "rph_agnonacimiento" "rph_edad" "rph_sexo"
## [34] "rph_idgen" "Kish" "rph_pertenencia_indigena"
## [37] "rph_nacionalidad" "rph_migracion" "rph_p9"
## [40] "rph_p10" "rph_p11" "rph_p12"
## [43] "rph_p13" "rph_p14" "Hora_termino_rph"
## [46] "P17" "P24" "A1_1_1"
## [49] "A1_1_1_N_Veces" "B1_1_1" "B1_1_1_N_Veces"
## [52] "C1_1_1" "C1_1_1_N_Veces" "D1_1_1"
## [55] "D1_1_1_N_Veces" "E1_1_1" "E1_1_1_N_Veces"
## [58] "F1_1_1" "G1_1_1" "G1_1_1_N_Veces"
## [61] "H1_1_1" "H1_1_1_N_Veces" "VA_DC"
## [64] "VP_DC" "DEN_AGREG" "RVA_DC"
## [67] "Hora_inicio_cc" "Hora_termino_cc" "Fact_Pers"
## [70] "Fact_Hog" "VarStrat" "Conglomerado"
## [73] "Fact_Ind"Es importante señalar que la base de datos se compone de variables de caracterización de los hogares y las personas (módulo de Registro de Personas en el Hogar o RPH) y variables temáticas de la encuesta referidas a percepción de inseguridad y victimización.
Adicionalmente, la base de datos contiene pesos muestrales, que deben ser considerados durante la anonimización. Para el nivel hogar se utiliza Fact_Hog, mientras que para el nivel persona se utiliza Fact_Ind.
Además, la base de datos cuenta con variables de Conglomerado y VarStrat, que serán utilizadas para declarar el diseño complejo y evaluar la utilidad de los datos antes y después del tratamiento de los mismos.
El archivo Diccionario de Variables.xlsx contiene la descripción de las variables contenidas en la base de datos.
11.1.2.3 Clasificación de variables
Las potenciales variables clave, que podría permitir una re-identificación, se ubican en el módulo de Registro de Personas en el Hogar (RPH), ya que permiten dar cuenta de atributos de las personas y hogares, como también en la portada de la encuesta, donde se encuentran las variables de ubicación geográfica. Estas últimas coinciden en parte con la base de datos de Hoja de Ruta, no obstante esta base no se publica.
11.1.2.4 Librerías requeridas
Se cargan las librerías requeridas para el proceso. En caso de que no estén instaladas, deberá instalarlas con la función install.packages() como se muestra en el siguiente ejemplo:
Bloque 11.4 Ejemplo de instalación de librería
install.packages("nombre_libreria") Luego, cargamos las librerías:
Bloque 11.5 Cargar librerías
library(sdcMicro) # paquete sdcMicro con funciones para el proceso SDC
library(survey) # para diseño complejo
library(calidad) # evaluación de calidad de las estimaciones
library(tidyverse) # herramientas para manipulación de datos
library(openxlsx) # lectura/escritura de archivos xlsx
library(stringr) # procesamiento de textos11.1.3 Determinación de necesidades de protección de confidencialidad
En esta actividad se describen el marco normativo y convenios a tener en cuenta para la anonimización, las unidades estadísticas contenidas en la base de datos, las variables sensibles contenidas en la base de datos, y el diagnóstico de necesidad de protección de confidencialidad.
11.1.3.1 Unidades estadísticas
Se verifica la cantidad de viviendas y de personas registradas en la base de datos. Para ello, se mide la cantidad de valores únicos del folio de viviendas (enc_idr) y el folio de personas (rph_ID).
Viviendas: 18.766 Personas: 47.344
Bloque 11.6 Contar la cantidad de folios a nivel de viviendas y personas
length(unique(file$enc_idr))## [1] 18766length(unique(file$rph_ID))## [1] 47344De este modo, se da cuenta de la cantidad de unidades estadísticas y de la estructura jerárquica de la base de datos.
11.1.3.2 variables sensibles
Se establecen como variables sensibles aquellas referidas a la tenencia de armas (P17), a elementos de seguridad en la vivienda (P24), y acerca de denuncia de delitos (DEN_AGREG).
Bloque 11.7 Guardar variables sensibles en un vector de texto
sensibles <- c('P17',
'P24',
'DEN_AGREG')Se verifica que las variables sensibles están presentes en la base de datos. Se espera que esta función devuelva el valor TRUE.
Bloque 11.8 Verificar presencia de variables sensibles
all(sensibles %in% names(file))## [1] TRUE11.1.4 Propiedades estadísticas a preservar
11.1.4.1 Usos claves de los datos
Se espera que los datos mantengan las siguientes propiedades:
Se debe poder reproducir los indicadores principales de la encuesta con precisión, por lo que idealmente no deben modificarse, con un máximo de diferencia en la estimación no mayor a un punto porcentual (diferencia en términos absolutos, dado que las estimaciones de ENUSC son de proporción).
Además, se deben mantener las variables de desagregación de tabulados de la encuesta, siendo estas sexo y región. Es decir, no deben observarse valores perdidos en estas variables.
Dado que las variables temáticas no corresponden a potenciales variables clave, se descuenta la posibilidad de modificar dichas variables, asegurando mantener sus propiedades estadísticas.
Por ende, el foco debe estar en mantener las relaciones entre las variables de desagregación que podrían ser modificadas como sexo y región, y las variables de los indicadores principales.
Un dato frecuentemente solicitado por transparencia es la variable comuna. Si bien el diseño muestral de ENUSC no permite realizar estimaciones a ese nivel de desagregación, esta variable es de todos modos de interés para los usuarios para análisis a nivel descriptivo y referencial. En este sentido, es pertinente evaluar la posibilidad de mantener esta variable en la base de datos anonimizada.
Por otro lado, la variable de edad en versiones anteriores de la ENUSC se publicaba con todos los valores de la variable (semi-continua), siendo ahora publicada como tramos etarios. Dado que usuarios del mundo académico y otros investigadores dan uso a esta variable, es también de interés analizar la posibilidad de mantenerla como semi-continua
11.1.4.2 Indicadores priorizados
Los indicadores priorizados corresponden a:
Victimización Agregada de Delitos Consumados Calculado en base a la variable VA_DC, por lo que la base de datos anonimizada debe permitir calcular este indicador a nivel nacional y regional (corresponde a hogares, por lo que no es desagregable por sexo).
Victimización Personal de Delitos Consumados Calculado en base a la variable VP_DC, por lo que la base de datos anonimizada debe permitir calcular este indicador a nivel nacional, regional y según sexo.
Finalmente, dado que la variable de edad es de interés para el análisis de cómo distintos grupos etarios se ven afectados por la delincuencia, es de interés que la relación entre la variable de edad y la variable de victimización personal se mantenga. Esto se evaluará a través de un modelo de regresión logística simple.
11.1.4.3 Medición de utilidad
11.1.4.3.1 Reproducción de estimaciones y desagregaciones
Dado que las variables de sexo, región y de los indicadores principales no serán modificadas se espera que estas mantengan las propiedades estadísticas de la base de datos original o no tratada con la mayor fidelidad posible. Partiendo del supuesto de que estas variables no serán modificadas, sino que el proceso SDC se aplicará sobre otras variables (principalmente otras variables del RPH), solo podría verse alteradas las estimaciones en caso de supresión de registros.
En este sentido, se espera que las estimaciones no difieran de la estimación original en más de un punto porcentual, en tanto diferencia en términos absolutos (todos los indicadores de ENUSC son de proporción).
A continuación, se presentan las estimaciones de la ENUSC utilizando el paquete de calidad de las estimaciones desarrollado en el INE, ya que también se espera que se mantengan los estándares de calidad de las estimaciones.
Ahora, se trabaja con un conjunto que contiene solo a los informantes que respondieron la encuesta (file_kish), descartando al resto de los integrantes del hogar. Esto es un paso necesario para poder declarar el diseño complejo de la encuesta.
Se establece el diseño complejo para personas y para hogares:
Bloque 11.9 Declarar el diseño complejo
file_kish <- file[file$Kish %in% 1,]
options(survey.lonely.psu = "certainty")
dc_pers <- svydesign(ids = ~Conglomerado,
strata = ~VarStrat,
data = file_kish,
weights = ~Fact_Pers)
dc_hog <- svydesign(ids = ~Conglomerado,
strata = ~VarStrat,
data = file_kish,
weights = ~Fact_Hog)Luego, realizamos las estimaciones desagregadas por región y sexo, guardando las tablas para posterior evaluación de la utilidad de los datos.
Victimización agregada de delitos consumados, desagregado por región:
Bloque 11.10 Estimar Victimización Agregada a nivel regional, con estandar de calidad INE
insumos_prop <- create_prop(var = 'VA_DC',
domains = 'enc_region',
design = dc_hog)
VA_DC_REG_PRE <- assess(insumos_prop)
VA_DC_REG_PRE[1:6]## enc_region stat se df n cv
## 1 1 0.5180939 0.01656239 1117 1283 0.03196793
## 2 2 0.4986086 0.02015781 701 834 0.04042813
## 3 3 0.5025207 0.01687235 1048 1224 0.03357544
## 4 4 0.5096128 0.01963187 763 892 0.03852312
## 5 5 0.5046148 0.01265927 1821 2106 0.02508700
## 6 6 0.4937284 0.01719661 1017 1169 0.03483011
## 7 7 0.5193102 0.01797112 920 1067 0.03460576
## 8 8 0.5170244 0.01427234 1474 1724 0.02760477
## 9 9 0.5070849 0.01951509 753 889 0.03848486
## 10 10 0.5261810 0.01672262 1085 1251 0.03178111
## 11 11 0.5240267 0.01849898 903 1066 0.03530160
## 12 12 0.5167737 0.01889486 796 932 0.03656313
## 13 13 0.5084057 0.02151621 604 731 0.04232094
## 14 14 0.5096388 0.01751652 975 1135 0.03437047
## 15 15 0.5026934 0.01590767 1191 1365 0.03164488
## 16 16 0.4899313 0.01751388 929 1098 0.03574763Victimización personal de delitos consumados, desagregado por sexo:
Bloque 11.11 Estimar Victimización Personal según sexo, con estandar de calidad INE
insumos_prop <- create_prop(var = 'VP_DC',
domains = 'rph_sexo',
design = dc_pers
)
VP_DC_SEXO_PRE <- assess(insumos_prop)
VP_DC_SEXO_PRE[1:6]## rph_sexo stat se df n cv
## 1 1 0.2526373 0.005327597 6721 9439 0.02108793
## 2 2 0.2447268 0.005269227 6590 9327 0.02153106Victimización personal de delitos consumados, desagregado por región:
Bloque 11.12 Estimar Victimización Personal a nivel regional, con estandar de calidad INE
insumos_prop <- create_prop(var = 'VP_DC',
domains = 'enc_region',
design = dc_pers)
VP_DC_REG_PRE <- assess(insumos_prop)
VP_DC_REG_PRE[1:6]## enc_region stat se df n cv
## 1 1 0.2568172 0.01420410 1117 1283 0.05530824
## 2 2 0.2313854 0.01700058 701 834 0.07347300
## 3 3 0.2379758 0.01447234 1048 1224 0.06081436
## 4 4 0.2475154 0.01704076 763 892 0.06884725
## 5 5 0.2422258 0.01087581 1821 2106 0.04489945
## 6 6 0.2521763 0.01503618 1017 1169 0.05962565
## 7 7 0.2685464 0.01612499 920 1067 0.06004546
## 8 8 0.2611829 0.01250193 1474 1724 0.04786657
## 9 9 0.2412502 0.01739434 753 889 0.07210085
## 10 10 0.2361271 0.01413669 1085 1251 0.05986900
## 11 11 0.2773473 0.01612302 903 1066 0.05813297
## 12 12 0.2396374 0.01618739 796 932 0.06754952
## 13 13 0.2462710 0.01884833 604 731 0.07653493
## 14 14 0.2527983 0.01529804 975 1135 0.06051481
## 15 15 0.2404411 0.01327941 1191 1365 0.05522937
## 16 16 0.2431671 0.01512454 929 1098 0.06219815Victimización personal de delitos consumados, desagregado por sexo y región:
Bloque 11.13 Estimar Victimización Personal a nivel regional y según sexo, con estandar de calidad INE
insumos_prop <- create_prop(var = 'VP_DC',
domains = 'rph_sexo+enc_region',
design = dc_pers)
VP_DC_REG_SEXO_PRE <- assess(insumos_prop)
VP_DC_REG_SEXO_PRE[1:6]## rph_sexo enc_region stat se df n
## 1 1 1 0.2612492 0.01993875 541 660
## 2 2 1 0.2522270 0.02020040 508 623
## 3 1 2 0.2138315 0.02274576 317 428
## 4 2 2 0.2493531 0.02527904 304 406
## 5 1 3 0.2392046 0.02068420 485 600
## 6 2 3 0.2367133 0.02041682 501 624
## 7 1 4 0.2571475 0.02439808 340 440
## 8 2 4 0.2388935 0.02367246 342 452
## 9 1 5 0.2582590 0.01563089 921 1067
## 10 2 5 0.2261775 0.01500918 889 1039
## 11 1 6 0.2557568 0.02147664 472 582
## 12 2 6 0.2486357 0.02088754 469 587
## 13 1 7 0.2751938 0.02324332 429 540
## 14 2 7 0.2616656 0.02226621 412 527
## 15 1 8 0.2826785 0.01822327 688 829
## 16 2 8 0.2408143 0.01715973 746 895
## 17 1 9 0.2479841 0.02381286 359 467
## 18 2 9 0.2333432 0.02543398 316 422
## 19 1 10 0.2458082 0.01995371 521 639
## 20 2 10 0.2262334 0.01988504 492 612
## 21 1 11 0.2660547 0.02254731 422 531
## 22 2 11 0.2894683 0.02341303 418 535
## 23 1 12 0.2383125 0.02254203 365 475
## 24 2 12 0.2410698 0.02307637 352 457
## 25 1 13 0.2515748 0.02693269 252 356
## 26 2 13 0.2411737 0.02646547 273 375
## 27 1 14 0.2533536 0.02087620 490 598
## 28 2 14 0.2521585 0.02282616 414 537
## 29 1 15 0.2408291 0.01870310 565 686
## 30 2 15 0.2400561 0.01891161 562 679
## 31 1 16 0.2262722 0.02063687 425 541
## 32 2 16 0.2592696 0.02167361 442 557Se espera que la base de datos anonimizada mantenga las propiedades aquí expuestas, por lo que se almacenan en objetos para su posterior comparación con los datos tratados.
Por otro lado, dado que queremos mantener la utilidad de los datos en relación con edad, se mide la relación entre la variable edad y la variable VP_DC, lo cual se debería mantener en caso que se deba recodificar la edad en tramos.
11.1.4.3.2 Relación entre variable de edad y victimización personal
Se genera un modelo logit con la variable de Victimización Personal como dependiente y con la variable de edad como regresor. Se espera que los resultados aquí obtenidos sean similares con los datos tratados, lo que se evaluará al final del proceso de anonimización.
Carga de datos
Primero, se cargan los datos, y se filtran dejando solo al informante Kish.
Bloque 11.14 Generar dataframe para modelo
data <- file[file$Kish %in% 1,]Dividir datos en training y testing sets
Luego, se dividen los datos en sets de training y testing.
Bloque 11.15 Dividir datos en training y testing sets
library(caTools)
set.seed(2022)
ids_train <- sample(data$rph_ID, nrow(data)/3*2)
training<- sjlabelled::remove_all_labels(data[data$rph_ID %in% ids_train,])
testing<- sjlabelled::remove_all_labels(data[!data$rph_ID %in% ids_train,])Construir modelo
Luego, construimos el modelo utilizando la función glm().
Bloque 11.16 Construir modelo
modelo<-glm(VP_DC~rph_edad,
data=training,
family = "binomial")
summary(modelo)##
## Call:
## glm(formula = VP_DC ~ rph_edad, family = "binomial", data = training)
##
## Deviance Residuals:
## Min 1Q Median 3Q Max
## -0.8025 -0.7661 -0.7320 -0.6980 1.7502
##
## Coefficients:
## Estimate Std. Error z value Pr(>|z|)
## (Intercept) -1.3401916 0.0525124 -25.52 < 2e-16 ***
## rph_edad 0.0034786 0.0007714 4.51 6.5e-06 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## (Dispersion parameter for binomial family taken to be 1)
##
## Null deviance: 13933 on 12509 degrees of freedom
## Residual deviance: 13912 on 12508 degrees of freedom
## AIC: 13916
##
## Number of Fisher Scoring iterations: 4Validación de modelo
Luego aplicamos el test de Hosmer Lemeshow para validar el modelo.
Bloque 11.17 Validación de modelo
library(ResourceSelection)
hoslem.test(modelo$y,fitted(modelo),g=10) # Test de Hosmer Lemeshow##
## Hosmer and Lemeshow goodness of fit (GOF) test
##
## data: modelo$y, fitted(modelo)
## X-squared = 255.13, df = 8, p-value < 2.2e-16library(pROC)
indiceC.trainig<-roc(modelo$y,fitted(modelo)) # Curva ROC## Setting levels: control = 0, case = 1## Setting direction: controls < casesindiceC.trainig##
## Call:
## roc.default(response = modelo$y, predictor = fitted(modelo))
##
## Data: fitted(modelo) in 9444 controls (modelo$y 0) < 3066 cases (modelo$y 1).
## Area under the curve: 0.5272Punto de corte óptimo
Se calcula el punto de corte óptimo utilizando la función coords().
Bloque 11.18 Calcular punto de corte óptimo
ptocorteop.training<-coords(indiceC.trainig,x="best",
input="threshold",
best.method="youden")
ptocorteop.training## threshold specificity sensitivity
## 1 0.270795 0.9431385 0.1457926library(ROCR)
ROC.training<-performance(prediction.obj = prediction(predictions = fitted(modelo),
labels = as.factor(modelo$y)),
"tpr",
"fpr")Luego, visualizamos el punto de corte óptimo utilizando la función plot().
Bloque 11.19 Visualizar punto de corte óptimo
plot(ROC.training, colorize=TRUE, print.cutoffs.at=seq(0.1, by=0.1))
abline(a=0,b=1)
abline(v=ptocorteop.training$threshold,col="red")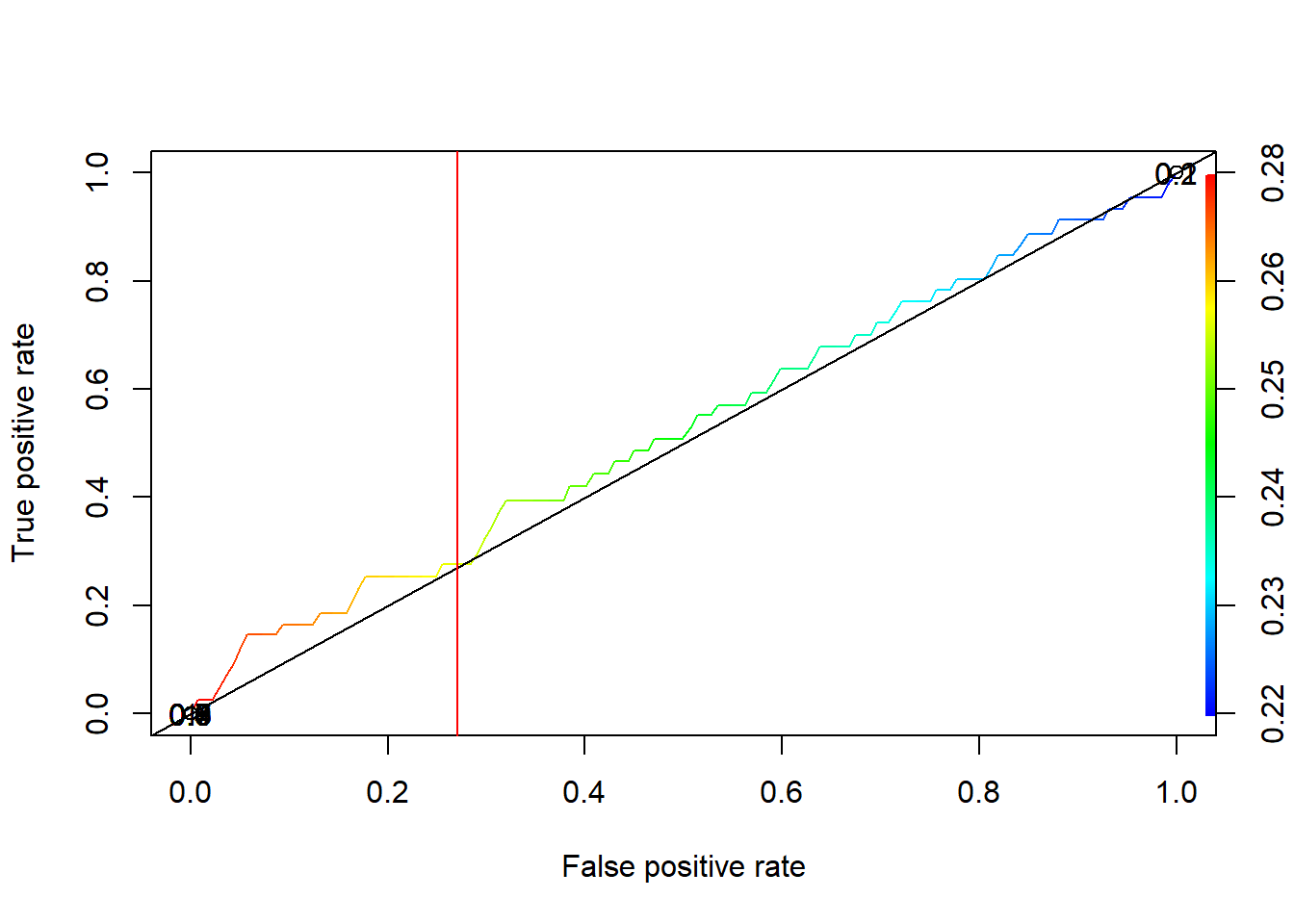
Figura 11.1: Punto de corte óptimo con datos no tratados
Predicciones y matriz de confusión
Se realizan predicciones y se genera una matriz de confusión.
Bloque 11.20 Predicciones y matriz de confusión
pred.training<-predict(modelo, data=training, type="response")
table(ActualValue=training$VP_DC,
PredictValue=pred.training>ptocorteop.training$threshold)## PredictValue
## ActualValue FALSE TRUE
## 0 8907 537
## 1 2619 447A continuación, se continua la evaluación del modelo con datos de prueba.
Validación de modelo (con datos de prueba)
Aplicamos nuevamente la validación con el test de Hosmer Lemeshow, esta vez con los datos de prueba.
Bloque 11.21 Validación de modelo con datos de prueba
hoslem.test(testing$VP_DC,predict(modelo,newdata=testing,type="response"),g=5) # Test de Hosmer Lemeshow##
## Hosmer and Lemeshow goodness of fit (GOF) test
##
## data: testing$VP_DC, predict(modelo, newdata = testing, type = "response")
## X-squared = 60.369, df = 3, p-value = 4.903e-13indiceC.testing=roc(testing$VP_DC,predict(modelo,newdata=testing,type="response")) # Curva ROC## Setting levels: control = 0, case = 1## Setting direction: controls < casesindiceC.testing##
## Call:
## roc.default(response = testing$VP_DC, predictor = predict(modelo, newdata = testing, type = "response"))
##
## Data: predict(modelo, newdata = testing, type = "response") in 4666 controls (testing$VP_DC 0) < 1590 cases (testing$VP_DC 1).
## Area under the curve: 0.5203Punto de corte óptimo (con datos de prueba)
Se calcula el punto de corte óptimo utilizando la función coords().
Bloque 11.22 Calcular de corte óptimo con datos de prueba
ptocorteop.testing<-coords(indiceC.testing,x="best",input="threshold",best.method="youden")
ptocorteop.testing## threshold specificity sensitivity
## 1 0.2633057 0.8317617 0.2440252ROC.testing<-performance(prediction(predict(modelo,newdata=testing,type="response"),
as.factor(testing$VP_DC)),"tpr","fpr")Luego, visualizamos el punto de corte óptimo utilizando la función plot().
Bloque 11.23 Visualizar de corte óptimo con datos de prueba
plot(ROC.testing, colorize=TRUE, print.cutoffs.at=seq(0.1, by=0.1))
abline(a=0,b=1)
abline(v=ptocorteop.testing$threshold,col="red")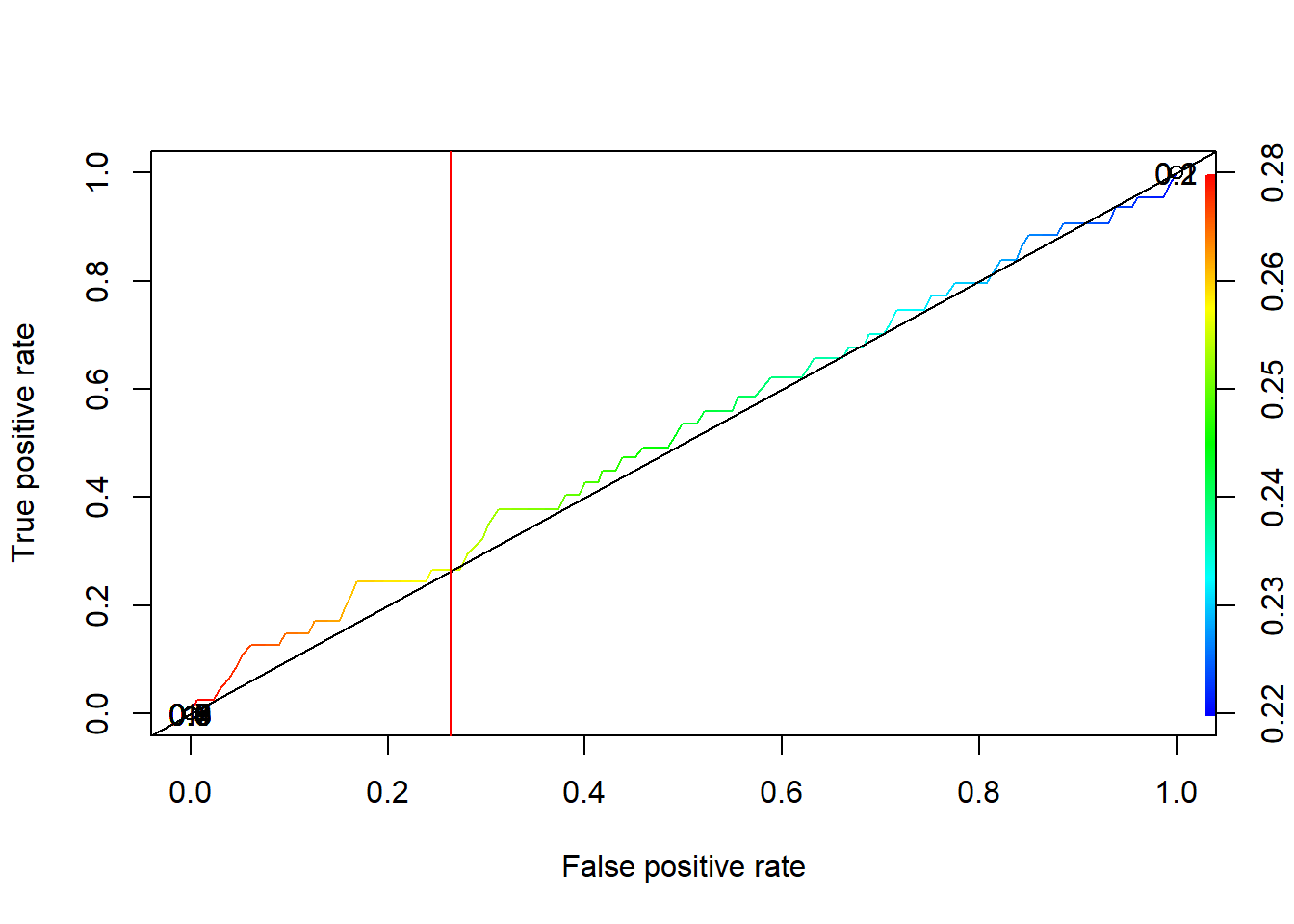
Figura 11.2: Punto de corte óptimo con datos de prueba a partir de datos no tratados
Predicciones y matriz de confusión (con datos de prueba)
Se realizan predicciones y se genera una matriz de confusión.
Bloque 11.24 Predicciones y matriz de confusión con datos de prueba
pred.testing<-predict(modelo, testing, type="response")
table(ActualValue=testing$VP_DC, PredictValue=pred.testing>ptocorteop.testing$threshold)## PredictValue
## ActualValue FALSE TRUE
## 0 3881 785
## 1 1202 388Comparar área bajo la curva, umbral, sensibilidad y especificidad
Finalmente, comparamos el área bajo la curva, umbral, sensibilidad y especificidad, a partir de los resultados del modelo con los datos de entrenamiento y con los datos de prueba.
Bloque 11.25 Comparar área bajo la curva, umbral, sensibilidad y especificidad
auc <-indiceC.trainig$auc - indiceC.testing$auc
corte <-ptocorteop.training$threshold - ptocorteop.testing$threshold
sens <- ptocorteop.training$sensitivity - ptocorteop.testing$sensitivity
spe <- ptocorteop.training$specificity - ptocorteop.testing$specificity11.2 Paso Dos: Preparar y explorar datos originales
11.2.1 Preparación de datos
11.2.1.1 Integración de datos
La base de datos ENUSC ya se encuentra integrada y no se combina con ninguna otra base producida para este producto estadístico.
11.2.1.2 Eliminación de identificadores directos
Se registra nombres de identificadores directos y otras variables internas del proyecto que no son publicadas.
Bloque 11.26 Almacenar indicadores directos y otras variables excluidas en un vector de texto
identificadores_directos <- c('enc_id','enc_distrito',
'enc_zona','enc_manzana','enc_vivienda','FECHA',
'enc_Nombre_ID',
'enc_Nombre_K','enc_Edad_K',
'rph_dianacimiento','rph_mesnacimiento',
'rph_agnonacimiento',
'rph_nombrepila','Hora_inicio_rph',
'Hora_termino_rph','Hora_inicio_cc','Hora_termino_cc',
'IDC','enc_letraKish',
'enc_direccion','enc_numero','enc_codfono',
'enc_fono','Enc_Fono_ID','Enc_Correo_ID',
'Enc_Fono_K','Enc_Correo_K')
all(identificadores_directos %in% names(file))## [1] TRUELuego, se quitan estas variables de la base de datos.
Bloque 11.27 Filtrar columnas excluidas
file <- file[,!names(file) %in% identificadores_directos]También, se quitan las variables de cadena con registros de observaciones, que tampoco corresponde publicar (en caso de que las hubiera). Estas corresponden a variables de texto que registran observaciones de terreno del encuestador y relatos de los delitos brindados por los informantes, información utilizada para el procesamiento de la base de datos, pero que no se consideran para su publicación.
Bloque 11.28 Filtrar columnas de texto y observaciones de terreno
file <- file[,!str_detect(names(file),'^Obs|Obs$')]11.2.1.3 Selección de Variables
En principio, todas las variables restantes son pertinentes de publicar, en la medida que se cumplan los requerimientos de anonimización de los datos.
Para efectos de los análisis siguientes y para la medición del riesgo, solo se considerará el siguiente listado de variables, que se considera que pueden ser utilizadas como variables clave para la re-identificación de los informantes. Todas estas corresponden a variables de ubicación o del RPH.
| Variable | Etiqueta |
|---|---|
| enc_rpc | Identificador de comuna |
| enc_region | Identificador de región |
| IH_residencia_habitual | Número de residentes habituales |
| rph_edad | Edad |
| rph_sexo | Sexo |
| rph_idgen | Identidad de Género |
| rph_pertenencia_indigena | Pertenencia a pueblos indígenas |
| rph_nacionalidad | Nacionalidad |
| rph_p14 | Razón para no buscar un empleo o iniciar una actividad por cuenta propia |
El resto de las variables que no son clave, pero se consideran para la publicación, se mantienen en el archivo de datos (variables temáticas sobre percepción de inseguridad y victimización).
11.2.1.4 Consolidación de variables
De las variables recién descritas, la variable comuna se encuentra anidada en la variable de región, por lo que hay redundancia al mantener ambas. Dado que comuna (enc_rpc) tiene mayor nivel de información, se utilizará primero esta variable para los siguientes análisis y la medición de riesgo, incluyendo región solo en caso de que sea necesario retirar la variable de comuna.
A continuación, se transforman y consolidan variables para poder aplicar adecuadamente los análisis.
Primero, las variables de comparte gastos y número de grupos se excluyen del análisis, ya que son redundantes con la cantidad de residentes habituales y cantidad de hogares, siendo utilizadas solo estas últimas dos.
Se fusionan categorías trans y otros, dado que son poco frecuentes.
Bloque 11.29 Consolidar categorías de identidad de género y sus valores perdidos
file$rph_idgen[file$rph_idgen %in% c(3,4)] <- 3
file$rph_idgen[file$rph_idgen %in% c(88,99,96)] <- NASe recodifican perdidos en diversas variables clave.
Bloque 11.30 Recodificar valores perdidos de pertenencia indígena
file$rph_pertenencia_indigena[file$rph_pertenencia_indigena %in% c(88,99,96)] <- NA
file$rph_nacionalidad[file$rph_nacionalidad %in% c(88,99,96)] <- NA
file$rph_p14[file$rph_p14 %in% c(88,99,96)] <- NAConsolidamos variable de situación ocupacional, ya que de otro modo tiene muchos valores perdidos por flujo, y apuntan a una sola clasificación con tres categorías que es lo más relevante.
Bloque 11.31 Consolidar situación ocupacional
# Generamos variable vacía
file$rph_situacion_ocupacional <- NA
# Ocupados
file$rph_situacion_ocupacional[file$rph_p9 %in% 1 | file$rph_p10 %in% 1] <- 1
# Desocupados
file$rph_situacion_ocupacional[file$rph_p12 %in% 1 & file$rph_p13 %in% 1] <- 2
# Inactivos
file$rph_situacion_ocupacional[file$rph_p12 %in% 2 | file$rph_p13 %in% 2] <- 3
# Revisamos
table(file$rph_situacion_ocupacional)##
## 1 2 3
## 30364 2470 7498Luego, integramos algunas categorías de inactivos con la condición de inactividad, ya que esto especifica subtipos de los inactivos que pueden ser relevantes para la re-identificación. Se agrupan las categorías que no son claves para re-identificar.
Bloque 11.32 Consolidar situación ocupacional con categorías de inactivos.
# Inactivo - Otros
file$rph_situacion_ocupacional[file$rph_situacion_ocupacional %in% 3 &
(file$rph_p14 %in% c(1,2,6,7) | is.na(file$rph_p14))] <- 3
# Inactivo - Estudiante
file$rph_situacion_ocupacional[file$rph_situacion_ocupacional %in% 3 &
file$rph_p14 %in% 3] <- 4
# Inactivo - Jubilado, pensionado o rentista
file$rph_situacion_ocupacional[file$rph_situacion_ocupacional %in% 3 &
file$rph_p14 %in% 4] <- 5
# Inactivo - Motivos de salud permanentes
file$rph_situacion_ocupacional[file$rph_situacion_ocupacional %in% 3 &
file$rph_p14 %in% 5] <- 6
# Revisamos
table(file$rph_situacion_ocupacional)##
## 1 2 3 4 5 6
## 30364 2470 5358 730 718 692Finalmente, se convierten las variables categóricas a tipo factor, quitándole el etiquetado de haven, ya que causa problemas con sdcMicro.
Bloque 11.33 Convertir variables clave a factor
file$enc_region <- as.factor(as.numeric(file$enc_region))
file$enc_rpc <- as.factor(as.numeric(file$enc_rpc))
file$rph_sexo <- as.factor(as.numeric(file$rph_sexo ))
file$rph_idgen <- as.factor(as.numeric(file$rph_idgen))
file$rph_pertenencia_indigena <- as.factor(as.numeric(file$rph_pertenencia_indigena))
file$rph_nacionalidad <- as.factor(as.numeric(file$rph_nacionalidad))
file$rph_situacion_ocupacional <- as.factor(as.numeric(file$rph_situacion_ocupacional))11.2.2 Exploración del conjunto de datos
Primero, revisamos cuáles variables tenemos en la base de datos en este punto del proceso.
Bloque 11.34 Revisar nuevamente variables presente en conjunto de datos
names(file)## [1] "enc_idr" "rph_ID" "enc_region"
## [4] "enc_rpc" "IH_residencia_habitual" "rph_numeroLinea"
## [7] "rph_parentesco" "rph_edad" "rph_sexo"
## [10] "rph_idgen" "Kish" "rph_pertenencia_indigena"
## [13] "rph_nacionalidad" "rph_migracion" "rph_p9"
## [16] "rph_p10" "rph_p11" "rph_p12"
## [19] "rph_p13" "rph_p14" "P17"
## [22] "P24" "A1_1_1" "A1_1_1_N_Veces"
## [25] "B1_1_1" "B1_1_1_N_Veces" "C1_1_1"
## [28] "C1_1_1_N_Veces" "D1_1_1" "D1_1_1_N_Veces"
## [31] "E1_1_1" "E1_1_1_N_Veces" "F1_1_1"
## [34] "G1_1_1" "G1_1_1_N_Veces" "H1_1_1"
## [37] "H1_1_1_N_Veces" "VA_DC" "VP_DC"
## [40] "DEN_AGREG" "RVA_DC" "Fact_Pers"
## [43] "Fact_Hog" "VarStrat" "Conglomerado"
## [46] "Fact_Ind" "rph_situacion_ocupacional"11.2.2.1 Cálculo de porcentaje de valores perdidos en las variables
Luego, calculamos el porcentaje de valores perdidos en las variables, considerando las celdas válidas según flujo de la encuesta (no se cuentan como valores perdidos las celdas vacías por saltos en el cuestionario). Aquellas variables que contengan más de un 50% de celdas perdidas, se excluyen del proceso de anonimización (pero sí deben incluirse en el archivo final de datos).
Se observa que no hay variables clave que tengan valores perdidos.
Bloque 11.35 Chequear valores perdidos
any(is.na(file$enc_region))## [1] FALSEany(is.na(file$enc_rpc))## [1] FALSEany(is.na(file$IH_residencia_habitual))## [1] FALSEany(is.na(file$rph_sexo))## [1] FALSEany(is.na(file$rph_edad))## [1] FALSEany(is.na(file$rph_pertenencia_indigena)) ## [1] FALSEany(is.na(file$rph_nacionalidad)) ## [1] FALSEany(is.na(file$rph_situacion_ocupacional[file$rph_edad > 14])) ## [1] FALSESe concluye que todas las variables cumplen con las condiciones para incluirse en el proceso de anonimización.
11.2.2.2 Cálculo de estadísticas de resumen
Por último, se revisan frecuencias para variables categóricas y estadísticos de resumen para numéricas. Esto permite visualizar que variables tienen categorías infrecuentes, lo que puede ser relevante más adelante para la toma de decisión de cuáles métodos aplicar y sobre cuáles variables.
Bloque 11.36 Tablas de frecuencia de variables clave
table(file$enc_region)##
## 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16
## 3223 2061 3115 2264 5269 2938 2648 4393 2241 3138 2693 2401 1842 2944 3368 2806table(file$enc_rpc)##
## 1101 1102 1103 1104 1105 1106 2101 2102 2103 2104 2105 2106 3101 3102 3103
## 561 601 522 500 533 506 326 355 350 358 343 329 524 564 493
## 3104 3105 3106 4101 4102 4103 4104 4105 4106 5101 5102 5103 5104 5105 5106
## 461 514 559 393 416 342 436 351 326 889 926 890 862 812 890
## 6101 6102 6103 6104 6105 6106 7101 7102 7103 7104 7105 7106 8101 8102 8103
## 495 471 455 507 522 488 434 470 395 463 409 477 698 720 774
## 8104 8105 8106 9101 9102 9103 9104 9105 9106 10101 10102 10103 10104 10105 10106
## 700 724 777 402 380 337 385 344 393 539 519 495 525 492 568
## 11101 11102 11103 11104 11105 11106 11107 12101 12102 12103 12104 12105 12106 12107 13101
## 340 350 380 446 375 432 370 322 302 382 411 340 362 282 251
## 13102 13103 13104 13105 13106 13107 14101 14102 14103 14104 14105 14106 14107 15101 15102
## 273 270 234 214 293 307 419 447 410 373 427 444 424 443 483
## 15103 15104 15105 15106 15107 16101 16102 16103 16104 16105 16106 16107
## 511 467 544 438 482 448 362 397 351 371 431 446table(file$rph_sexo)##
## 1 2
## 23694 23650table(file$rph_idgen)##
## 1 2 3
## 23679 23615 50table(file$rph_pertenencia_indigena)##
## 1 2 3 4 5 6 7 8 9 10 11
## 49 59 51 49 51 35 64 44 50 48 46844table(file$rph_nacionalidad)##
## 1 2 3 4 5 6 7 8 9
## 38923 1049 1010 1060 1025 1048 1035 1125 1069table(file$rph_situacion_ocupacional)##
## 1 2 3 4 5 6
## 30364 2470 5358 730 718 692Bloque 11.37 Estadísticos de resumen de variables clave
summary(file$IH_residencia_habitual)## Min. 1st Qu. Median Mean 3rd Qu. Max.
## 1.000 2.000 3.000 2.942 4.000 4.000summary(file$rph_edad)## Min. 1st Qu. Median Mean 3rd Qu. Max.
## 0.00 25.00 53.00 53.06 80.00 107.0011.3 Paso Tres: Medición y evaluación del riesgo de divulgación
11.3.1 Definición de escenarios de divulgación
Las variables escogidas en su gran mayoría se encuentran contenidas también en otros productos estadísticos del INE y otras entidades públicas, ya que refieren a variables básicas de ubicación y caracterización sociodemográfica. Se destaca la coincidencia de variables de caracterización de personas de los hogares con CASEN y EPF, como ejemplos de fuentes internas. En el caso de fuentes externas, la ENPG de SENDA se considera una encuesta que contiene variables similares para hacer match.
Por este motivo, se toma un único escenario conservador, en que todas estas variables son potencialmente utilizables en combinación por un intruso, siendo posibles de enlazar a otras base de datos públicas y/o privadas, habilitando una posible re-identificación de registros.
En este sentido, y considerando las variables ya fusionadas/consolidadas, se mantiene el siguiente listado de variables para la medición del riesgo:
| Variable | Etiqueta |
|---|---|
| enc_rpc | Identificador de comuna |
| enc_region | Identificador de región |
| IH_residencia_habitual | Número de residentes habituales |
| rph_edad | Edad |
| rph_sexo | Sexo |
| rph_idgen | Identidad de género |
| rph_pertenencia_indigena | Pertenencia a pueblos indígenas |
| rph_nacionalidad | Nacionalidad |
| rph_situacion_ocupacional | Situación ocupacional |
11.3.2 Medición de riesgos de divulgación
Para medir el riesgo se considerarán las medidas de riesgo global, riesgo individual, l-diversity y k-anonimato,considerando además la estructura jerárquica de la base. Por este motivo, primero se mide el riesgo a nivel de vivienda/hogar, para luego medir a nivel individual.
11.3.2.1 Medición de riesgos de divulgación a nivel vivienda/hogar
En los siguientes pasos, se seleccionan las variables del nivel jerárquico superior y se genera el objeto sdcMicro en base al cual se realizan las estimaciones.
Primero, se generan vectores con las variables claves y las variables numéricas en el conjunto de datos (nivel hogar). Luego, opcionalmente se pueden declarar variables para el método PRAM (en este caso se genera un vector vacío ya que no se utilizará el método PRAM).
Bloque 11.38 Generar vectores con variables claves categóricas, numéricas y variables PRAM
# Selección de variables para la anonimización a nivel de hogar
selectedKeyVarsHH <- c("enc_rpc")
# variables numericas
numVarsHH <- c("IH_residencia_habitual")
# No se declaran variables para método PRAM
pramVarsHH <- c()A continuación, se genera un vector con la variable de ponderación de hogares.
Bloque 11.39 Asignar variables de ponderación a nivel hogar
# Se indican las variables de ponderación de hogares
weightVarsHH <- c("Fact_Hog")Ahora, reunimos todas estas variables en un vector denominado HHVars
Bloque 11.40 Generar vector con variables del nivel hogar
# Luego se genera un vector con todas las variables del nivel de hogar
HHVars <- c('enc_idr',selectedKeyVarsHH, pramVarsHH, numVarsHH, weightVarsHH)
variables <- HHVars
todas_variables <- names(file)
HHVars <- intersect(todas_variables,variables)Luego, generamos un conjunto de datos con solo las columnas y filas que corresponden. En este caso son las variables del nivel hogar y las filas que corresponden al informante Kish, de manera tal que haya un caso por hogar.
Bloque 11.41 Filtrar un caso por vivienda
# Creamos un subconjunto de datos de file con hogares y variables HH
fileHH <- file[,HHVars]
fileHH$Kish <- file$Kish
# Se deja un caso por cada hogar asignado en fileHH
fileHH <- fileHH[fileHH$Kish %in% 1,]
fileHH <- dplyr::select(fileHH, -Kish)Luego, se verifican las dimensiones del conjunto de datos con la función dim().
Bloque 11.42 Construir e inspeccionar dataframe de hogares
# Se genera el dataset para medición de riesgo
fileHH <- data.frame(enc_idr = fileHH$enc_idr,
enc_rpc = fileHH$enc_rpc,
IH_residencia_habitual = fileHH$IH_residencia_habitual,
Fact_Hog = fileHH$Fact_Hog)
# Se verifican dimensiones del conjunto de datos
dim(fileHH)## [1] 18766 4Y se genera el objeto SDC a nivel de hogar.
Bloque 11.43 Crear objeto SDC
# Se crea objeto SDC inicial para variables de nivel de hogar
sdcHH <- createSdcObj(dat = fileHH, keyVars = selectedKeyVarsHH,
numVars = numVarsHH, weightVar = weightVarsHH)Por último, se guarda en un vector la cantidad de hogares para uso posterior.
Bloque 11.44 Almacenar número de hogares
# Se genera variable con número de hogares
numHH <- length(fileHH[,1]) # número de hogares Primero, revisamos las medidas globales de riesgo. Se observa que no hay hasta el momento observaciones que tengan un riesgo superior que la mayoría de los datos.
Bloque 11.45 Medidas globales de riesgo
print(sdcHH, "risk")## Risk measures:
##
## Number of observations with higher risk than the main part of the data: 0
## Expected number of re-identifications: 0.16 (0.00 %)Ahora medimos el riesgo de manera individual. Se observan 0 casos con riesgo sobre el 1%.
Bloque 11.46 Riesgo individual
# Observations with risk above certain threshold (0.01)
nrow(fileHH[sdcHH@risk$individual[, "risk"] > 0.01,])## [1] 0Revisamos el k-anonimato para los casos ponderados.No hay casos que violen ningún nivel de k-anonimato. Para ello, se usa la función kAnon_violations(), indicando TRUE (T) como argumento para el parámetro de pesos (weighted) y el valor de k a evaluar.
Bloque 11.47 K-anonimato
print(sdcHH)## Infos on 2/3-Anonymity:
##
## Number of observations violating
## - 2-anonymity: 0 (0.000%)
## - 3-anonymity: 0 (0.000%)
## - 5-anonymity: 0 (0.000%)
##
## ----------------------------------------------------------------------En resumen, se observa que para el nivel hogar los riesgos ya cumplen con los umbrales establecidos por el estándar de anonimización. Por lo que se prosigue analizando el riesgo a nivel de personas.
11.3.2.2 Medición de riesgos de divulgación a nivel persona
En este siguiente paso, se procede a seleccionar todas las variables de ambos niveles, para crear el objeto sdcMicro considerando la estructura jerárquica hasta el nivel persona.
Bloque 11.48 Comandos para construir el objeto SDC a nivel de persona
# Se indican variables clave (nivel individual)
selectedKeyVarsIND <- c('enc_rpc',
'rph_edad',
'rph_sexo',
'rph_idgen',
'rph_pertenencia_indigena',
'rph_nacionalidad',
'rph_situacion_ocupacional')
# Se indica factor de expansión de personas
WeightVarIND <- c('Fact_Ind')
# ID Hogares
selectedHouseholdID <- c('enc_idr')
# Recombinación de conjuntos de datos HH anónimos y variables de nivel individuales
indVars <- c("enc_idr", "rph_ID", selectedKeyVarsIND,WeightVarIND,sensibles) # HID and all non HH variables
fileInd <- file[indVars] # subset of file without HHVars
fileCombined <- dplyr::inner_join(fileInd, select(fileHH, -enc_rpc), by= c('enc_idr'))
dim(fileCombined)## [1] 47344 15# Objetos SDC con todas las variables y variables HH tratadas para
# anonimización de variables de nivel individual
sdcCombined <- createSdcObj(dat = fileCombined, keyVars = selectedKeyVarsIND,
hhId = selectedHouseholdID, weightVar = WeightVarIND,
sensibleVar = sensibles)Primero, revisamos las medidas globales de riesgo. Se observa que existen casos de observaciones con riesgo alto, y también un porcentaje de re-identificaciones esperadas, lo que se acentúa al considerar la estructura jerárquica. No obstante, a nivel global, esta no supera el umbral establecido de no más del 10%.
Bloque 11.49 Medidas globales de riesgo
print(sdcCombined, "risk")## Risk measures:
##
## Number of observations with higher risk than the main part of the data: 1530
## Expected number of re-identifications: 1117.28 (2.36 %)
##
## Information on hierarchical risk:
## Expected number of re-identifications: 3113.28 (6.58 %)
## ----------------------------------------------------------------------Ahora medimos el riesgo de manera individual. Para ello, se revisa la cantidad de observaciones con riesgo mayor a cada umbral de riesgo individual. Se observa que casi todos los casos presentan un riesgo superior al 1%, lo que incumple el umbral establecido. Al filtrar por porcentajes de riesgo más alto, se observa aún una cantidad muy alta de observaciones con riesgo altísimo, con 141 observaciones con riesgo superior al 50%.
Bloque 11.50 Riesgo individual
# Observaciones con riesgo individual superior al 1%:
nrow(fileCombined[sdcCombined@risk$individual[, "risk"] > 0.01,])## [1] 22797# Observaciones con riesgo individual superior al 5%:
nrow(fileCombined[sdcCombined@risk$individual[, "risk"] > 0.05,]) ## [1] 4405# Observaciones con riesgo individual superior al 25%:
nrow(fileCombined[sdcCombined@risk$individual[, "risk"] > 0.25,]) ## [1] 409# Observaciones con riesgo individual superior al 50%:
nrow(fileCombined[sdcCombined@risk$individual[, "risk"] > 0.5,]) ## [1] 141Luego, revisamos el k-anonimato para los casos ponderados. Se detectan casi la mitad de los casos incumplen el 2-anonimato a nivel muestral.
Bloque 11.51 K-anonimato
print(sdcCombined)## Infos on 2/3-Anonymity:
##
## Number of observations violating
## - 2-anonymity: 22806 (48.171%)
## - 3-anonymity: 33996 (71.806%)
## - 5-anonymity: 44657 (94.325%)
##
## ----------------------------------------------------------------------Finalmente, medimos el l-diversity, que es una medida complementaria al k-anonimato. Esta indica cuantos valores de respuesta tienen las variables sensibles para cada combinación de las variables clave. Se espera obtener valores superiores a 1, dado que este valor indica que hay una única respuesta para cada combinación, lo que implica que un intruso podría saber el valor de respuesta a pesar de que se cumplan los umbrales de k-anonimato. En este sentido, esta métrica de riesgo es complementaria y da respaldo a lo evaluado a partir del k-anonimato.
Bloque 11.52 L-Diversity
# Generamos el objeto de l-diversity
med_riesgo <- ldiversity(sdcCombined, ldiv_index = sensibles,
l_recurs_c = 2, missing = NA)
# revisamos las medidas de riesgo de l-diversity
med_riesgo@risk$ldiversity## --------------------------## L-Diversity Measures## --------------------------## P17_Distinct_Ldiversity P24_Distinct_Ldiversity DEN_AGREG_Distinct_Ldiversity
## Min. :1.000 Min. :1.000 Min. :1.000
## 1st Qu.:1.000 1st Qu.:1.000 1st Qu.:1.000
## Median :1.000 Median :1.000 Median :1.000
## Mean :1.561 Mean :1.651 Mean :1.591
## 3rd Qu.:2.000 3rd Qu.:2.000 3rd Qu.:2.000
## Max. :7.000 Max. :8.000 Max. :7.000Como se señaló previamente, el valor 1 indica un mínimo nivel de diversidad en las variables sensibles para cada combinación de variables clave. En este sentido, el que la media y promedio del l-diversity se aproximen a 1 nos indica que tenemos pocas combinaciones, lo que es señal de un mayor riesgo de que el intruso logre llegar al valor de las variables sensibles.
11.3.3 Evaluación de riesgos de divulgación
CONCLUSIÓN DE LA EVALUACIÓN DE RIESGOS
Dado que, a nivel jerárquico, considerando hasta el nivel de persona, se incumplen los umbrales requeridos por el estándar de anonimización a nivel individual y de k-anonimato, además de que se observa un valor riesgoso en el l-diversity, se confirma la necesidad de aplicar métodos SDC para asegurar la confidencialidad de los datos.
Estos métodos se continúan aplicando sobre el nivel de personas del conjunto de datos, dado que a nivel hogares ya se cumplen los umbrales requeridos.
11.4 Paso Cuatro: Selección y aplicación de métodos SDC
En esta sección se aplicarán iterativamente métodos SDC intentando alcanzar los umbrales de riesgo requeridos. Como veremos, al ser un proceso iterativo, también considerará de forma recurrente la re-medición del riesgo, que corresponde en estricto rigor a la primera parte del paso cinco. Esto es necesario para ir evaluando si es necesario aplicar métodos adicionales o distintos.
11.4.1 Primer conjunto de métodos SDC
El primer método a aplicar es la recodificación global para la variable de edad,pasando de semi continua a ordinal. Los tramos etarios escogidos corresponden a una adaptación más desagregada de los tramos etarios utilizados en la publicación de tabulados en versiones anteriores de la encuesta.
Se procede con este método primero dado que, por experiencia de los analistas de la encuesta, es uno de los que tiene mayor impacto en la reducción de riesgos.
Para ello, aplicamos la función globalRecode(). Esta función implementa el método de recodificación global descrito en la guía de anonimización. El método toma cuatro argumentos: el objeto SDC, la columna a recodificar, los límites de los intervalos a generar, y las etiquetas que debe asignar a cada tramo.
Bloque 11.53 Recodificar edad
sdcCombined <- globalRecode(sdcCombined,
column = "rph_edad",
breaks=c(-1,14,19,24,29,39,49,59,69,79,89,120),
labels=0:10)Alternativamente, se podría simplemente haber ocupado otras funciones de R base o de Tidyverse para recodificar esta variable en tramos, como, por ejemplo, las funciones ifelse() o case_when(). Sin embargo, esta aproximación tiene la desventaja de que requiere generar nuevamente el objeto SDC por completo, por lo que se pierde la trazabilidad de las ediciones realizadas y de la reducción del riesgo desde la línea base inicial. Por este motivo, es que se recomienda usar globalRecode().
Luego de haber aplicado este método SDC, procedemos a medir el impacto de esta modificación en las medidas de riesgo.
11.4.2 Re-medición del riesgo para primer conjunto de métodos
Primero, revisamos las medidas globales de riesgo con los datos tratados. Se observa una mejora en los resultados, llegando nuevamente a umbrales aceptables (<10%). No obstante, es necesario revisar el resto de las métricas, donde se está más lejos de cumplir con los estándares requeridos.
Bloque 11.54 Riesgo global
print(sdcCombined, "risk")## Risk measures:
##
## Number of observations with higher risk than the main part of the data:
## in modified data: 612
## in original data: 1530
## Expected number of re-identifications:
## in modified data: 435.53 (0.92 %)
## in original data: 1117.28 (2.36 %)
##
## Information on hierarchical risk:
## Expected number of re-identifications:
## in modified data: 1300.72 (2.75 %)
## in original data: 3113.28 (6.58 %)
## ----------------------------------------------------------------------Ahora medimos el riesgo de manera individual. Se observa que 8.759 de los casos tiene un riesgo superior al 1%. Al filtrar por porcentajes de riesgo más alto, se observa aún una cantidad menor de observaciones con riesgo altísimo, con 52 observaciones con riesgo superior al 50%, lo que aún es demasiado alto.
Bloque 11.55 Riesgo individual
# Observaciones con riesgo individual superior al 1%:
nrow(fileCombined[sdcCombined@risk$individual[, "risk"] > 0.01,])## [1] 8759# Observaciones con riesgo individual superior al 5%:
nrow(fileCombined[sdcCombined@risk$individual[, "risk"] > 0.05,])## [1] 1713# Observaciones con riesgo individual superior al 25%:
nrow(fileCombined[sdcCombined@risk$individual[, "risk"] > 0.25,])## [1] 178# Observaciones con riesgo individual superior al 50%:
nrow(fileCombined[sdcCombined@risk$individual[, "risk"] > 0.5,])## [1] 52Revisamos el k-anonimato para los casos a nivel muestral. Aún se observa un muy alto porcentaje de casos que incumplen el 2-anonimato.
Bloque 11.56 K-anonimato
print(sdcCombined)## Infos on 2/3-Anonymity:
##
## Number of observations violating
## - 2-anonymity: 8936 (18.875%) | in original data: 22806 (48.171%)
## - 3-anonymity: 12778 (26.990%) | in original data: 33996 (71.806%)
## - 5-anonymity: 16183 (34.182%) | in original data: 44657 (94.325%)
##
## ----------------------------------------------------------------------Luego medimos el l-diversity, donde observamos mejores valores que se alejan de 1, para las tres variables sensibles.
Bloque 11.57 L-diveristy
# Generamos el objeto de l-diversity
med_riesgo <- ldiversity(sdcCombined, ldiv_index = sensibles,
l_recurs_c = 2, missing = NA)
# revisamos las medidas de riesgo de l-diversity
med_riesgo@risk$ldiversity## --------------------------## L-Diversity Measures## --------------------------## P17_Distinct_Ldiversity P24_Distinct_Ldiversity DEN_AGREG_Distinct_Ldiversity
## Min. : 1.000 Min. : 1.000 Min. : 1.000
## 1st Qu.: 1.000 1st Qu.: 1.000 1st Qu.: 1.000
## Median : 4.000 Median : 5.000 Median : 4.000
## Mean : 5.851 Mean : 6.327 Mean : 6.093
## 3rd Qu.: 9.000 3rd Qu.:10.000 3rd Qu.:10.000
## Max. :35.000 Max. :36.000 Max. :36.000En conclusión, si bien se cumplen los umbrales a nivel global, los umbrales a nivel individual y el k-anonimato nos indican que se requiere aplicar más métodos SDC para asegurar la anonimización de los datos.
11.4.3 Segundo conjunto de métodos SDC
El hecho de estar aplicando nuevamente el paso 4 nos indica una característica fundamental de los procesos de anonimización, que es que estos son procesos iterativos, donde es necesario ir aplicando métodos progresivamente y monitorear los niveles de riesgo en cada iteración. Como veremos en este ejercicio aplicado, serán varias iteraciones antes de llegar a un resultado satisfactorio de niveles de riesgo.
Retomando el proceso, el segundo método a aplicar es la eliminación de la variable de comuna. En cambio, se incluye la variable de región, ya que deja de ser redundante. Esto, dado que previamente se había retirado en el paso de consolidación de variables puesto que la comuna es una variable anidada en la variable de región.
Los pasos aplicados en el siguiente bloque de código son los mismos que se aplicaron la primera vez que se construyó el objeto sdcCombined. La única diferencia es que se reemplaza la variable enc_rpc por enc_region dentro del vector de variables clave selectedKeyVarsIND.
Bloque 11.58 Reconstruir el objeto SDC a nivel persona con variable región
# Se indican variables clave (nivel individual)
selectedKeyVarsIND <- c('enc_region',
'rph_edad',
'rph_sexo',
'rph_idgen',
'rph_pertenencia_indigena',
'rph_nacionalidad',
'rph_situacion_ocupacional')
# Se indica factor de expansión de personas
WeightVarIND <- c('Fact_Ind')
# ID Hogares
selectedHouseholdID <- c('enc_idr')
# Recombinación de conjuntos de datos HH anónimos y variables de nivel individuales
indVars <- c("enc_idr", "rph_ID", selectedKeyVarsIND,WeightVarIND,sensibles) # HID and all non HH variables
fileInd <- file[indVars] # subset of file without HHVars
fileCombined <- dplyr::inner_join(fileInd, select(fileHH, -enc_rpc), by= c('enc_idr'))
dim(fileCombined)## [1] 47344 15# Objetos SDC con todas las variables y variables HH tratadas para
# anonimización de variables de nivel individual
sdcCombined <- createSdcObj(dat = fileCombined, keyVars = selectedKeyVarsIND,
hhId = selectedHouseholdID, weightVar = WeightVarIND,
sensibleVar = sensibles)Además, como en este caso fue necesario crear nuevamente el objeto SDC, hay que aplicar nuevamente la recodificación de la variable edad con la función globalRecode().
Bloque 11.59 Recodificar edad nuevamente
sdcCombined <- globalRecode(sdcCombined,
column = "rph_edad",
breaks=c(-1,14,19,24,29,39,49,59,69,79,89,120),
labels=0:10)11.4.4 Re-medición del riesgo para segundo conjunto de métodos
Nuevamente, revisamos las medidas globales de riesgo. Se observa que a nivel global obtenemos valores aceptables de riesgo, ya que se habían logrado en la primera iteración.
Bloque 11.60 Riesgo global
print(sdcCombined, "risk")## Risk measures:
##
## Number of observations with higher risk than the main part of the data:
## in modified data: 178
## in original data: 712
## Expected number of re-identifications:
## in modified data: 147.03 (0.31 %)
## in original data: 529.44 (1.12 %)
##
## Information on hierarchical risk:
## Expected number of re-identifications:
## in modified data: 446.46 (0.94 %)
## in original data: 1565.30 (3.31 %)
## ----------------------------------------------------------------------Ahora medimos el riesgo de manera individual. Se observa que 2.902 de los casos presentan un riesgo superior al 1%, lo que aún cumple el umbral establecido, que indica el 20% de los casos. Por otro lado, se observa que 517 de las personas presentan un riesgo mayor al 5%, lo que cumple con el umbral (no más de 15%). Por otro lado, se observa que 50 y un 17 de los casos tienen riesgos superiores al 25% y 50%, respectivamente. Esto incumple con los umbrales establecidos ya que se espera que no haya observaciones con estos niveles de riesgo.
Bloque 11.61 Riesgo individual
# Observaciones con riesgo individual superior al 1%:
nrow(fileCombined[sdcCombined@risk$individual[, "risk"] > 0.01,])## [1] 2902# Observaciones con riesgo individual superior al 5%:
nrow(fileCombined[sdcCombined@risk$individual[, "risk"] > 0.05,])## [1] 517# Observaciones con riesgo individual superior al 25%:
nrow(fileCombined[sdcCombined@risk$individual[, "risk"] > 0.25,])## [1] 50# Observaciones con riesgo individual superior al 50%:
nrow(fileCombined[sdcCombined@risk$individual[, "risk"] > 0.5,])## [1] 17Revisamos el k-anonimato para los casos no ponderados, es decir a nivel muestral. Aún hay 2.886 casos que incumplen el 2-anonimato.
Bloque 11.62 K-anonimato
print(sdcCombined)## Infos on 2/3-Anonymity:
##
## Number of observations violating
## - 2-anonymity: 2886 (6.096%) | in original data: 10798 (22.808%)
## - 3-anonymity: 4994 (10.548%) | in original data: 14650 (30.944%)
## - 5-anonymity: 8341 (17.618%) | in original data: 18033 (38.089%)
##
## ----------------------------------------------------------------------Luego medimos el l-diversity. Se observa una importante mejora en el l-diversity, al observarse ahora una diversidad de 30 en promedio para las distintas combinaciones de variables clave en relación con las variables sensibles.
Bloque 11.63 L-diversity
# Generamos el objeto de l-diversity
med_riesgo <- ldiversity(sdcCombined, ldiv_index = sensibles,
l_recurs_c = 2, missing = NA)
# revisamos las medidas de riesgo de l-diversity
med_riesgo@risk$ldiversity## --------------------------## L-Diversity Measures## --------------------------## P17_Distinct_Ldiversity P24_Distinct_Ldiversity DEN_AGREG_Distinct_Ldiversity
## Min. : 1.00 Min. : 1.00 Min. : 1.00
## 1st Qu.: 2.00 1st Qu.: 2.00 1st Qu.: 2.00
## Median : 21.00 Median : 22.00 Median : 21.00
## Mean : 29.58 Mean : 30.09 Mean : 30.01
## 3rd Qu.: 47.00 3rd Qu.: 48.00 3rd Qu.: 48.00
## Max. :154.00 Max. :155.00 Max. :155.00En suma, debido a que persiste cierto grado de riesgos individuales e incumplimiento de k-anonimato aún se requiere seguir aplicando métodos SDC.
11.4.5 Tercer conjunto de métodos SDC
Como tercera iteración, se aplican varios métodos para poder reducir los riesgos individuales y el incumplimiento de 2-anonimato, que son las métricas que han presentado mayor dificultad para disminuir.
Primero, se recodifican globalmente las variables de pertenencia indígena y nacionalidad. Para esto se ocupa una función distinta, groupAndRename(), que permite también recodificar variables categóricas. También se fusionan categorías de situación laboral, lo que equivale a posteriormente eliminar la variable de razón de inactividad de la base de datos. Esto porque esta variable corresponde a una consolidación de variables (revisar paso 2.1.4).
Bloque 11.64 Recodificar de variables
# Recodificamos pertenencia indígena
sdcCombined <- groupAndRename(sdcCombined, var="rph_pertenencia_indigena",
before=c(1:10), after=c(1))
sdcCombined <- groupAndRename(sdcCombined, var="rph_pertenencia_indigena",
before=c(11), after=c(2))
# Recodificamos nacionalidad
sdcCombined <- groupAndRename(sdcCombined, var="rph_nacionalidad",
before=c(2:9), after=c(2))
# Recodificamos situacion_ocupacional
sdcCombined <- groupAndRename(sdcCombined, var="rph_situacion_ocupacional",
before=c(3:6), after=c(3))Luego, se aplica supresión local en las variables de menor prioridad. Se seleccionan estas variables cuidadosamente, dado que se aplicará un método drástico como es la supresión local, teniendo como criterio las propiedades estadísticas que se buscan preservar en la base de datos.
Por otro lado, los umbrales que se entregan como argumento a esta función en el parámetro threshold dependen de la data y se debe probar iterativamente hasta lograr los umbrales deseados. Se debe introducir en los argumentos umbrales de riesgo no tan bajos, evitando sobre-anonimizar, dado que esto nos llevaría a perder más utilidad de la necesaria. En este caso, se indican umbrales de 0.1, es decir, del 10% de riesgo individual como objetivo.
Bloque 11.65 Supresión local
# supresión local para riesgos globales e individuales
sdcCombined <- localSupp(sdcCombined, keyVar='rph_idgen', threshold=0.01)
sdcCombined <- localSupp(sdcCombined, keyVar='rph_pertenencia_indigena', threshold=0.01)
sdcCombined <- localSupp(sdcCombined, keyVar='rph_nacionalidad', threshold=0.01)
sdcCombined <- localSupp(sdcCombined, keyVar='rph_situacion_ocupacional', threshold=0.01)
sdcCombined <- localSuppression(sdcCombined,importance = 1:7, k = 2)Como veremos en el siguiente paso,los métodos aplicados en esta tercera iteración logran alcanzar los umbrales requeridos. Con esto, podemos pasar al Paso Cinco y evaluar de forma completa el proceso SDC, consirando tanto la evaluación del riesgo como de la utilidad.
11.5 Paso Cinco: Evaluar proceso SDC
11.5.1 Re-medición del riesgo
Primero, revisamos las medidas globales de riesgo. Se observa, valores mucho menores de riesgo global, aun cuando esto ya cumplía previamente con los niveles esperados.
Bloque 11.66 Riesgo global
print(sdcCombined, "risk")## Risk measures:
##
## Number of observations with higher risk than the main part of the data:
## in modified data: 0
## in original data: 712
## Expected number of re-identifications:
## in modified data: 9.15 (0.02 %)
## in original data: 529.44 (1.12 %)
##
## Information on hierarchical risk:
## Expected number of re-identifications:
## in modified data: 26.98 (0.06 %)
## in original data: 1565.30 (3.31 %)
## ----------------------------------------------------------------------Ahora medimos el riesgo de manera individual. Se observa que no hay casos que excedan estos niveles de riesgo luego de los últimos tratamientos a los datos.
Bloque 11.67 Riesgo individual
# Observaciones con riesgo individual superior al 1%:
nrow(fileCombined[sdcCombined@risk$individual[, "risk"] > 0.01,])## [1] 0# Observaciones con riesgo individual superior al 5%:
nrow(fileCombined[sdcCombined@risk$individual[, "risk"] > 0.05,])## [1] 0# Observaciones con riesgo individual superior al 25%:
nrow(fileCombined[sdcCombined@risk$individual[, "risk"] > 0.25,])## [1] 0# Observaciones con riesgo individual superior al 50%:
nrow(fileCombined[sdcCombined@risk$individual[, "risk"] > 0.5,])## [1] 0Revisamos el k-anonimato para los casos ponderados. Ahora, no hay casos que violen ningún nivel de 2-anonimato y una cantidad aceptable de casos que incumplen el 3-anonimato y 5-anonimato, por lo que se cumpliría de buena forma lo planteado para estos umbrales.
Bloque 11.68 K-anonimato
print(sdcCombined)## Infos on 2/3-Anonymity:
##
## Number of observations violating
## - 2-anonymity: 0 (0.000%) | in original data: 10798 (22.808%)
## - 3-anonymity: 367 (0.775%) | in original data: 14650 (30.944%)
## - 5-anonymity: 1281 (2.706%) | in original data: 18033 (38.089%)
##
## ----------------------------------------------------------------------Luego medimos el l-diversity. Se observa que los valores de promedio y mediana de l-diversity son levemente superiores, lo que indican un menor nivel de riesgo de que el intruso logre acertar a los valores de las variables sensibles. Dado que se obtienen buenos resultados, se decide no proseguir con métodos SDC para no perder más utilidad de los datos.
Bloque 11.69 L-diversity
# Generamos el objeto de l-diversity
med_riesgo <- ldiversity(sdcCombined, ldiv_index = sensibles,
l_recurs_c = 2, missing = NA)
# revisamos las medidas de riesgo de l-diversity
med_riesgo@risk$ldiversity## --------------------------## L-Diversity Measures## --------------------------## P17_Distinct_Ldiversity P24_Distinct_Ldiversity DEN_AGREG_Distinct_Ldiversity
## Min. : 1.00 Min. : 1.00 Min. : 1.00
## 1st Qu.: 7.00 1st Qu.: 7.00 1st Qu.: 7.00
## Median : 23.00 Median : 24.00 Median : 24.00
## Mean : 31.64 Mean : 32.19 Mean : 32.11
## 3rd Qu.: 47.00 3rd Qu.: 48.00 3rd Qu.: 48.00
## Max. :154.00 Max. :155.00 Max. :155.00Los resultados aquí expuestos cumplen con los umbrales, por lo que ahora corresponde volver a medir la utilidad.
11.5.2 Evaluar proceso SDC - Volver a medir utilidad
11.5.2.1 Extracción de datos tratados y medición de perdida de información.
Primero, extraemos la data tratada. Para ello se usa la función extractManipData().
Bloque 11.70 Extraer datos tratados
fileTratada <- extractManipData(sdcCombined)Luego evaluamos cuántos valores perdidos tenemos debido a la supresión local. Para ello, imprimimos el porcentaje de celdas perdidas pre y post anonimización.
Primero vemos que, por flujo algunas variables tienen celdas vacías. La comparación consiste en ver si luego estos porcentajes aumentan.
Bloque 11.71 Celdas vacías previo a anomización
# % de celdas vacías pre-anonimización
for(i in 1:length(names(fileCombined))){
print(names(fileCombined)[i])
print(sum(is.na(fileCombined[[names(fileCombined)[i]]]))/nrow(fileCombined)*100)
}## [1] "enc_idr"
## [1] 0
## [1] "rph_ID"
## [1] 0
## [1] "enc_region"
## [1] 0
## [1] "rph_edad"
## [1] 0
## [1] "rph_sexo"
## [1] 0
## [1] "rph_idgen"
## [1] 0
## [1] "rph_pertenencia_indigena"
## [1] 0
## [1] "rph_nacionalidad"
## [1] 0
## [1] "rph_situacion_ocupacional"
## [1] 14.81075
## [1] "Fact_Ind"
## [1] 0
## [1] "P17"
## [1] 60.36245
## [1] "P24"
## [1] 60.36245
## [1] "DEN_AGREG"
## [1] 60.36245
## [1] "IH_residencia_habitual"
## [1] 0
## [1] "Fact_Hog"
## [1] 0Acá vemos los porcentajes de valores perdidos en los datos tratados. Vemos que en las variables de identidad de género (rph_idgen), pertenencia indígena (rph_pertenencia_indigena) y nacionalidad (rph_nacionalidad), hay un leve aumento de celdas en blanco. Esto es muy marginal, siendo menos de 2% de celdas vacías adicionales en identidad de género y menos de 1% adicional de celdas vacías en las otras dos variables. Además, se observa que para el caso de situación ocupacional (rph_situacion_ocupacional), el algoritmo de supresión local no eliminó valores, dado que los umbrales ya se habían cumplido.
Bloque 11.72 Celdas vacías después de anomización
for(i in 1:length(names(fileTratada))){
print(names(fileTratada)[i])
print(sum(is.na(fileTratada[[names(fileTratada)[i]]]))/nrow(fileTratada)*100)
}## [1] "enc_idr"
## [1] 0
## [1] "rph_ID"
## [1] 0
## [1] "enc_region"
## [1] 0
## [1] "rph_edad"
## [1] 0
## [1] "rph_sexo"
## [1] 0
## [1] "rph_idgen"
## [1] 1.203954
## [1] "rph_pertenencia_indigena"
## [1] 0.9314802
## [1] "rph_nacionalidad"
## [1] 0.3527374
## [1] "rph_situacion_ocupacional"
## [1] 14.872
## [1] "Fact_Ind"
## [1] 0
## [1] "P17"
## [1] 60.36245
## [1] "P24"
## [1] 60.36245
## [1] "DEN_AGREG"
## [1] 60.36245
## [1] "IH_residencia_habitual"
## [1] 0
## [1] "Fact_Hog"
## [1] 0En el resto de las variables, no hay pérdida de información.
11.5.2.2 Propiedades estadísticas priorizadas
Ahora, se procede a evaluar si se mantienen las propiedades estadísticas priorizadas de los datos.
Primero, se genera una variable de edad a partir de la marca de clase de los tramos etarios, para evaluar si se mantiene la relación con los indicadores principales. Esto no se evaluará inmediatamente, pero se genera para uso posterior. Es importante aclarar que esta recodificación se realiza solo con fines analíticos para determinar la utilidad de los datos anonimizados, no siendo utilizada para los datos a liberar (en estos se mantienen los tramos etarios).
Bloque 11.73 Generar variable de marcas de clase de edad
fileTratada$rph_edad_mc <- dplyr::case_when(
fileTratada$rph_edad == 1 ~ 7.5,
fileTratada$rph_edad == 2 ~ 17,
fileTratada$rph_edad == 3 ~ 22,
fileTratada$rph_edad == 4 ~ 27,
fileTratada$rph_edad == 5 ~ 34.5,
fileTratada$rph_edad == 6 ~ 44.5,
fileTratada$rph_edad == 7 ~ 54.5,
fileTratada$rph_edad == 8 ~ 64.5,
fileTratada$rph_edad == 9 ~ 74.5,
fileTratada$rph_edad == 10 ~ 84.5,
fileTratada$rph_edad == 11 ~ 90
)
fileTratada$rph_edad_mc <- as.numeric(fileTratada$rph_edad_mc)
table(fileTratada$rph_edad_mc)##
## 7.5 17 22 27 34.5 44.5 54.5 64.5 74.5 84.5 90
## 7012 2216 2228 2114 4324 4282 4274 4304 4397 4348 7845Luego, se pegan las variables de diseño muestral e indicadores a evaluar, ya que no se encuentran dentro del conjunto de datos con que se trabajó durante el proceso de anonimización.
Bloque 11.74 Añadir variables de diseño muestral
fileTratada <- dplyr::left_join(fileTratada,
file[,c('rph_ID','Fact_Pers','Fact_Hog',
'Conglomerado','VarStrat',
'VA_DC','VP_DC','Kish')])## Joining with `by = join_by(rph_ID, Fact_Hog)`A continuación, se comparan los resultados de los datos originales con los tratados en lo que refiere al cálculo de indicadores principales con desagregaciones.
Para ello, primero se establece el diseño complejo para personas y para hogares.
Bloque 11.75 Declarar diseño complejo
# generamos un conjunto de datos tratados donde solo tenemos las filas del informante Kish
fileTratada_kish <- fileTratada[fileTratada$Kish %in% 1,]
# fijamos las opciones del diseño complejo
options(survey.lonely.psu = "certainty")
# generamos el diseño complejo para el factor de expansión de personas
dc_pers_trat <- svydesign(ids = ~Conglomerado,
strata = ~VarStrat,
data = fileTratada_kish,
weights = ~Fact_Pers)
# generamos el diseño complejo para el factor de expansión de hogares
dc_hog_trat <- svydesign(ids = ~Conglomerado,
strata = ~VarStrat,
data = fileTratada_kish,
weights = ~Fact_Hog)Previamente, calculamos estas desagregaciones con los datos no tratados. A continuación, se recalculan con los datos tratados y se comparan los resultados calculando la diferencia. Esta se visualiza a través de la función summary().
Victimización agregada de delitos consumados, desagregado por región:
Bloque 11.76 Estimar Victimización Agregada a nivel regional, con estandar de calidad INE
insumos_prop <- create_prop(var = 'VA_DC',
domains = 'enc_region',
design = dc_hog_trat)
VA_DC_REG_TRAT <- assess(insumos_prop)
summary(VA_DC_REG_PRE$objetivo - VA_DC_REG_TRAT$objetivo)## Min. 1st Qu. Median Mean 3rd Qu. Max.
## Victimización personal de delitos consumados, desagregado por sexo:
Bloque 11.77 Estimar Victimización Personal según sexo, con estandar de calidad INE
insumos_prop <- create_prop(var = 'VP_DC',
domains = 'rph_sexo',
design = dc_pers_trat)
VP_DC_SEXO_TRAT <- assess(insumos_prop)
summary(VP_DC_SEXO_PRE$objetivo - VP_DC_SEXO_TRAT$objetivo)## Min. 1st Qu. Median Mean 3rd Qu. Max.
## Victimización personal de delitos consumados, desagregado por región:
Bloque 11.78 Estimar Victimización Personal a nivel regional, con estandar de calidad INE
insumos_prop <- create_prop(var = 'VP_DC',
domains = 'enc_region',
design = dc_pers_trat)
VP_DC_REG_TRAT <- assess(insumos_prop)
summary(VP_DC_REG_PRE$objetivo - VP_DC_REG_TRAT$objetivo)## Min. 1st Qu. Median Mean 3rd Qu. Max.
## Victimización personal de delitos consumados, desagregado por sexo y región:
Bloque 11.79 Estimar Victimización Personal a nivel regional y según sexo, con estandar de calidad INE
insumos_prop <- create_prop(var = 'VP_DC',
domains = 'rph_sexo+enc_region',
design = dc_pers_trat)
VP_DC_REG_SEXO_TRAT <- assess(insumos_prop)
summary(VP_DC_REG_SEXO_PRE$objetivo - VP_DC_REG_SEXO_TRAT$objetivo)## Min. 1st Qu. Median Mean 3rd Qu. Max.
## En resumen, se cumple para todos los casos que no hay diferencias en las estimaciones al calcular con los datos originales y los tratados.
Por último, se evalúa si se mantiene la relación entre la variable de edad y el indicador de Victimización Personal, utilizando las marcas de clase de los tramos etarios presentes en los datos tratados.
Si bien, los modelos obtenidos no son buenos predictores de la victimización, ya que es un fenómeno influenciado por múltiples factores y acá solo se está considerando la edad, si se aprecia que los resultados son similares a los de los datos sin tratar. En particular el coeficiente beta de la variable de edad en el modelo logit de victimización personal es prácticamente el mismo que en los datos no tratados.
Se genera un dataframe “data” para trabajar con el modelo
Primero, se cargan los datos, y se filtran dejando solo al informante Kish.
Bloque 11.80 Generar dataframe para modelo
data <- fileTratada_kishDividir datos en _training_ and _testing_ sets
Luego, se dividen los datos en sets de training y testing.
Bloque 11.81 Dividir datos en training y testing sets
library(caTools)
set.seed(2022)
ids_train <- sample(data$rph_ID, nrow(data)/3*2)
training<- sjlabelled::remove_all_labels(data[data$rph_ID %in% ids_train,])
testing<- sjlabelled::remove_all_labels(data[!data$rph_ID %in% ids_train,])Construir modelo
Luego, construimos el modelo utilizando la función glm().
Bloque 11.82 Construir modelo
modelo<-glm(VP_DC~rph_edad_mc,
data=training,
family = "binomial")
summary(modelo)##
## Call:
## glm(formula = VP_DC ~ rph_edad_mc, family = "binomial", data = training)
##
## Deviance Residuals:
## Min 1Q Median 3Q Max
## -0.7857 -0.7670 -0.7317 -0.7004 1.7468
##
## Coefficients:
## Estimate Std. Error z value Pr(>|z|)
## (Intercept) -1.3416201 0.0556848 -24.093 < 2e-16 ***
## rph_edad_mc 0.0036047 0.0008517 4.232 2.31e-05 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## (Dispersion parameter for binomial family taken to be 1)
##
## Null deviance: 13933 on 12509 degrees of freedom
## Residual deviance: 13915 on 12508 degrees of freedom
## AIC: 13919
##
## Number of Fisher Scoring iterations: 4Validación de modelo
Luego aplicamos el test de Hosmer Lemeshow para validar el modelo.
Bloque 11.83 Validación de modelo
library(ResourceSelection)
hoslem.test(modelo$y,fitted(modelo),g=10) # Test de Hosmer Lemeshow##
## Hosmer and Lemeshow goodness of fit (GOF) test
##
## data: modelo$y, fitted(modelo)
## X-squared = 156.47, df = 6, p-value < 2.2e-16library(pROC)
indiceC.trainig<-roc(modelo$y,fitted(modelo)) # Curva ROC## Setting levels: control = 0, case = 1## Setting direction: controls < casesindiceC.trainig##
## Call:
## roc.default(response = modelo$y, predictor = fitted(modelo))
##
## Data: fitted(modelo) in 9444 controls (modelo$y 0) < 3066 cases (modelo$y 1).
## Area under the curve: 0.5302Punto de corte óptimo
Se calcula el punto de corte óptimo utilizando la función coords().
Bloque 11.84 Calcular punto de corte óptimo
ptocorteop.training<-coords(indiceC.trainig,x="best",
input="threshold",
best.method="youden")
ptocorteop.training## threshold specificity sensitivity
## 1 0.2636494 0.8235917 0.2524462library(ROCR)
ROC.training<-performance(prediction.obj = prediction(predictions = fitted(modelo),
labels = as.factor(modelo$y)),
"tpr",
"fpr")Luego, visualizamos el punto de corte óptimo utilizando la función plot().
Bloque 11.85 Visualizar punto de corte óptimo
plot(ROC.training, colorize=TRUE, print.cutoffs.at=seq(0.1, by=0.1))
abline(a=0,b=1)
abline(v=ptocorteop.training$threshold,col="red")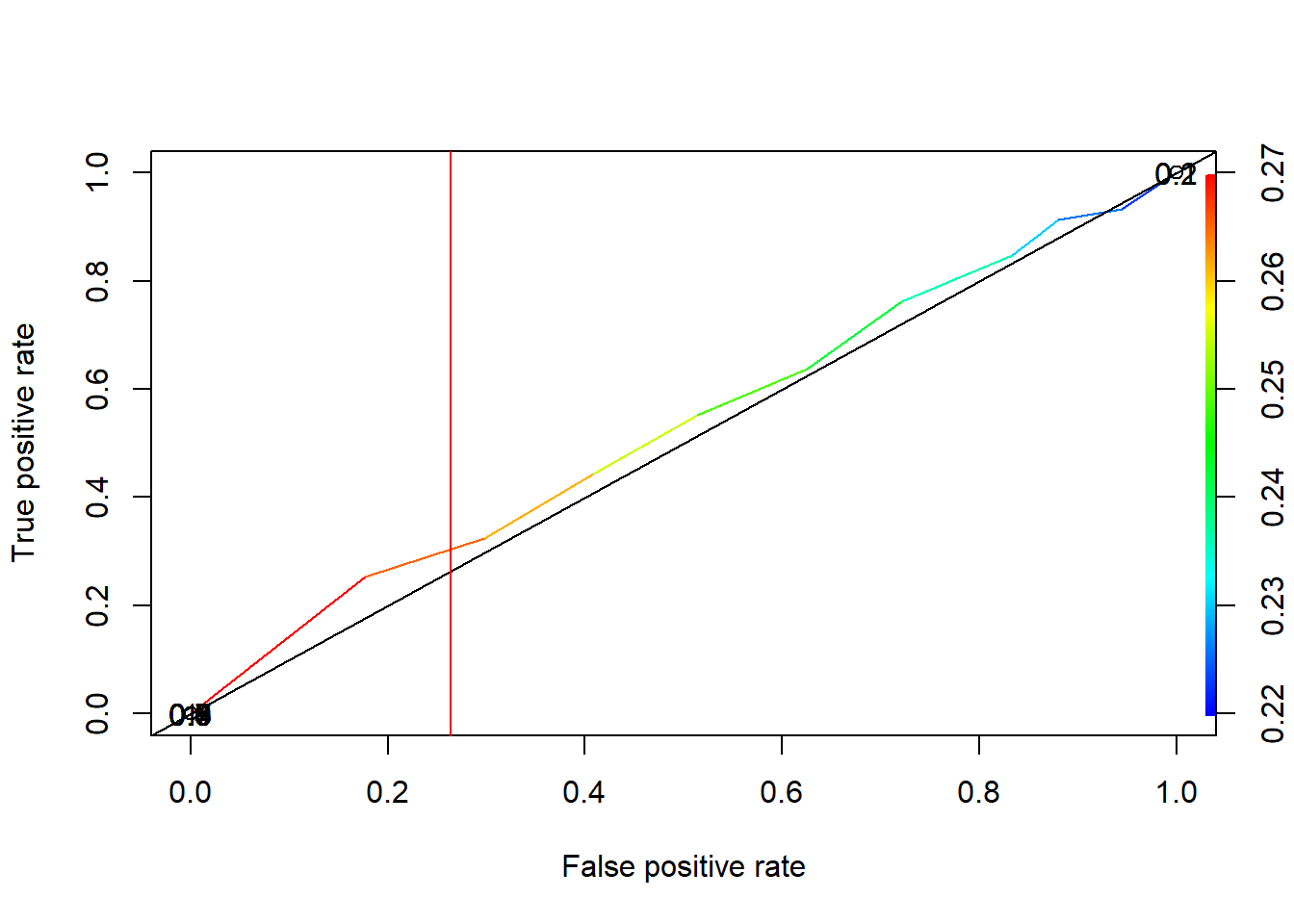
Figura 11.3: Punto de corte óptimo con datos tratados
Predicciones y matriz de confusión
Se realizan predicciones y se genera una matriz de confusión.
Bloque 11.86 Predicciones y matriz de confusión
pred.training<-predict(modelo, data=training, type="response")
table(ActualValue=training$VP_DC,
PredictValue=pred.training>ptocorteop.training$threshold)## PredictValue
## ActualValue FALSE TRUE
## 0 7778 1666
## 1 2292 774A continuación, se continua la evaluación del modelo con datos de prueba.
Validación de modelo (con datos de prueba)
Aplicamos nuevamente la validación con el test de Hosmer Lemeshow, esta vez con los datos de prueba.
Bloque 11.87 Validación de modelo con datos de prueba
hoslem.test(testing$VP_DC,predict(modelo,newdata=testing,type="response"),g=5) # Test de Hosmer Lemeshow##
## Hosmer and Lemeshow goodness of fit (GOF) test
##
## data: testing$VP_DC, predict(modelo, newdata = testing, type = "response")
## X-squared = 66.467, df = 3, p-value = 2.431e-14indiceC.testing=roc(testing$VP_DC,predict(modelo,newdata=testing,type="response")) # Curva ROC## Setting levels: control = 0, case = 1## Setting direction: controls < casesindiceC.testing##
## Call:
## roc.default(response = testing$VP_DC, predictor = predict(modelo, newdata = testing, type = "response"))
##
## Data: predict(modelo, newdata = testing, type = "response") in 4666 controls (testing$VP_DC 0) < 1590 cases (testing$VP_DC 1).
## Area under the curve: 0.5243Punto de corte óptimo (con datos de prueba)
Se calcula el punto de corte óptimo utilizando la función coords().
Bloque 11.88 Calcular de corte óptimo con datos de prueba
ptocorteop.testing<-coords(indiceC.testing,x="best",input="threshold",best.method="youden")
ptocorteop.testing## threshold specificity sensitivity
## 1 0.2636494 0.8317617 0.2440252ROC.testing<-performance(prediction(predict(modelo,newdata=testing,type="response"),
as.factor(testing$VP_DC)),"tpr","fpr")Luego, visualizamos el punto de corte óptimo utilizando la función plot().
Bloque 11.89 Visualizar de corte óptimo con datos de prueba
plot(ROC.testing, colorize=TRUE, print.cutoffs.at=seq(0.1, by=0.1))
abline(a=0,b=1)
abline(v=ptocorteop.testing$threshold,col="red")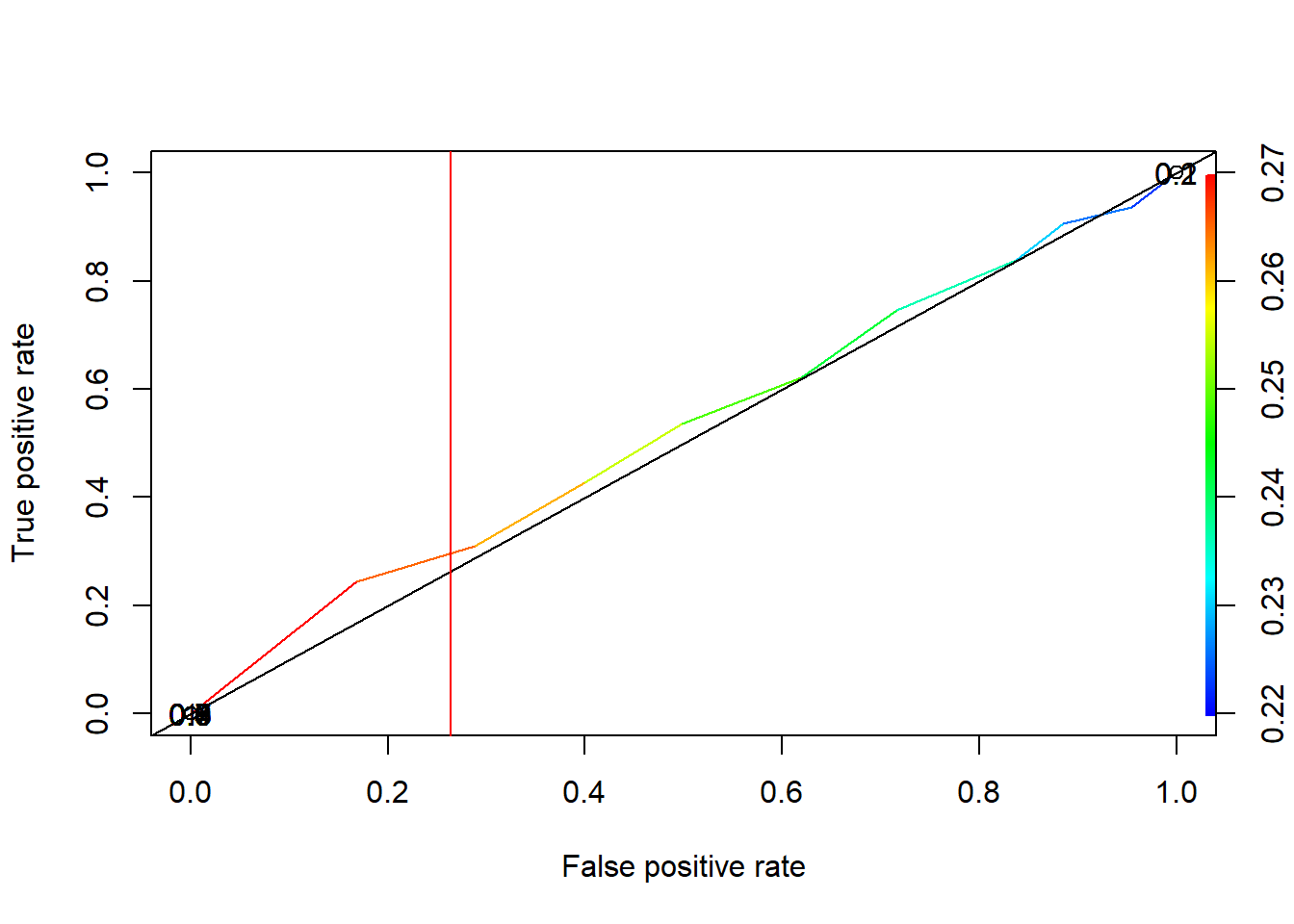
Figura 11.4: Punto de corte óptimo con datos de prueba a partir de datos tratados
Predicciones y matriz de confusión (con datos de prueba)
Se realizan predicciones y se genera una matriz de confusión.
Bloque 11.90 Predicciones y matriz de confusión con datos de prueba
pred.testing<-predict(modelo, testing, type="response")
table(ActualValue=testing$VP_DC, PredictValue=pred.testing>ptocorteop.testing$threshold)## PredictValue
## ActualValue FALSE TRUE
## 0 3881 785
## 1 1202 388Compara área bajo la curva, umbral, sensibilidad y especificidad
Finalmente, comparamos el área bajo la curva, umbral, sensibilidad y especificidad, a partir de los resultados del modelo con los datos de entrenamiento y con los datos de prueba.
Bloque 11.91 Comparar área bajo la curva, umbral, sensibilidad y especificidad
auc <-indiceC.trainig$auc - indiceC.testing$auc
corte <-ptocorteop.training$threshold - ptocorteop.testing$threshold
sens <- ptocorteop.training$sensitivity - ptocorteop.testing$sensitivity
spe <- ptocorteop.training$specificity - ptocorteop.testing$specificityLa siguiente tabla muestra las estimaciones del coeficiente beta de la variable independiente de edad en los modelos, para los datos originales y los datos tratados. Se observa que ambas estimaciones son equivalentes, sin diferencias estadísticamente significativas a partir de su intervalo de confianza:
| Datos | Estimación | Lim. Inf. | Lim. Sup. |
|---|---|---|---|
| Originales | 0,0035 | 0,0020 | 0,0050 |
| Tratados | 0,0036 | 0,0019 | 0,0053 |
En suma, se observa que se mantienen las propiedades estadísticas priorizadas de la base de datos, por lo que cumple con las condiciones para su liberación.
11.6 Paso Seis: Generar Reportes y Liberar Datos
11.6.1 Reportes
Al realizar un ejercicio de anonimización como el recién expuesto, se debe elaborar un reporte que documente el proceso de anonimización, con sus antecedentes y resultados. Para ello, debe basarse en el modelo de reporte de anonimización descrito en la guía de anonimización.
11.6.2 Liberación de datos
A partir del objeto file, que son los datos originales, se genera un objeto data.frame para su exportación como archivo de datos que será liberado.
Primero se verifica que el orden de registros sea idéntico entra datos originales y tratados. Esto permite sobre-escribir columnas completas, sabiendo que los registros coincidirán bien. En este caso, es importante usar el folio que corresponde a persona, ya que cada fila es una persona (en contraposición al folio de viviendas que cubre varias filas dependiendo de las personas que las componen).
Bloque 11.92 Verificar coincidencia de folios
# Se espera que esta función regrese TRUE
all(file$rph_ID == fileTratada$rph_ID)## [1] TRUELuego, se reemplazan variables tratadas en datos originales y se eliminan las variables que están de más. Estas son, comuna y razón de inactividad, ya que esta última permitiría deshacer la recodificación que se realizó en la variable consolidada de situación ocupacional.
Bloque 11.93 Eliminar variables
file$enc_rpc <- NULL
file$rph_p14 <- NULLLuego, se reemplazan variables tratadas de edad, identidad de género, pertenencia indígena y nacionalidad.
Bloque 11.94 Reemplazar variables originales por variables tratadas
file$rph_edad <- fileTratada$rph_edad
file$rph_idgen <- fileTratada$rph_idgen
file$rph_pertenencia_indigena <- fileTratada$rph_pertenencia_indigena
file$rph_nacionalidad <- fileTratada$rph_nacionalidadSe eliminan los valores de rph_p9 a rph_p13 para los casos suprimidos en situación ocupacional
Bloque 11.95 Suprimir valores
# se eliminan los valores que corresponden
file$rph_p9[is.na(fileTratada$rph_situacion_ocupacional)] <- NA
file$rph_p10[is.na(fileTratada$rph_situacion_ocupacional)] <- NA
file$rph_p11[is.na(fileTratada$rph_situacion_ocupacional)] <- NA
file$rph_p12[is.na(fileTratada$rph_situacion_ocupacional)] <- NA
file$rph_p13[is.na(fileTratada$rph_situacion_ocupacional)] <- NA
# Luego eliminamos situación ocupacional ya que no se usará más
file$rph_situacion_ocupacional <- NULLLuego, se elimina los valores de rph_migración que son acompañados de un NA en nacionalidad, ya que sería un error de flujo de la encuesta mantenerlos.
Bloque 11.96 Suprimir valores en nacionalidad
file$rph_migracion[is.na(file$rph_nacionalidad)] <- NAPor último, se exporta archivo de datos ocupando la librería haven.
Bloque 11.97 Ejemplo de código para exportar datos anonimizados
#haven::write_sav(file, 'ENUSC2020_anonimizada.sav')11.6.3 Anexo : Tablas de indicadores con desagregaciones
Se deja como anexo los tabulados con datos tratados y no tratados por si se considera necesario compararlos de forma manual.
Bloque 11.98 Ejemplo de código para exportar datos de indicadores con datos tratados y no tratados
#anexo <- list('P1 Regional Original' = P1_REG_PRE,
# 'P1 Regional Tratado' = P1_REG_TRAT,
# 'P1 Sexo Original' = P1_SEXO_PRE,
# 'P1 Sexo Tratado' = P1_SEXO_TRAT,
# 'P1 Regional Sexo Original' = P1_REG_SEXO_PRE,
# 'P1 Regional Sexo Tratado' = P1_REG_SEXO_TRAT,
# 'Vict. Pers. Regional Original' = VP_DC_REG_PRE,
# 'Vict. Pers. Regional Tratado' = VP_DC_REG_TRAT,
# 'Vict. Pers. Sexo Original' = VP_DC_SEXO_PRE,
# 'Vict. Pers. Sexo Tratado' = VP_DC_SEXO_TRAT,
# 'Vict. Pers. Regional Sexo Original' = VP_DC_REG_SEXO_PRE,
# 'Vict. Pers. Regional Sexo Tratado' = VP_DC_REG_SEXO_TRAT,
# 'Vict. Agr. Regional Original' = VA_DC_REG_PRE,
# 'Vict. Agr. Regional Tratado' = VA_DC_REG_TRAT)
#
#openxlsx::write.xlsx(anexo, "Anexo_Medicion_Utilidad.xlsx")